library(tidyverse)
library(janitor)
library(nortest)
library(DataExplorer)
library(ggrepel)
library (plotly)
library(MASS)
library(rgl)
library(car)
library(nlme)Regressão Linear
Base de Dados
Para os próximos exemplos iremos utilizar a base de dados MTCARS que possui informações sobre 32 veículos de revista Motor Trends de 1974.
# Selecionar base mtcars e criar uma coluna com os nomes dos modelos
df <- mtcars |>
rownames_to_column(var = "name") |> as_tibble()
dfVariável dependente
Para nossos experimentos, iremos selecionar como variável dependente o consumo dos veículos por distância rodada (milhas por galão ou mpg). Ou seja, tentaremos criar modelo preditivo que, com base em variáveis explicativas presentes em nossa amostra de dados, tentarão prever o valor do consumo (mpg) de um automóvel, que é uma variável quantitativa.
Visualizando a variável dependente
Apenas para começar, vamos dar uma breve olhada em nossa variável dependente:
summary(mtcars$mpg) Min. 1st Qu. Median Mean 3rd Qu. Max.
10.40 15.43 19.20 20.09 22.80 33.90 Aqui, vemos que temos um consumo mínimo de 10.4 e máximo de 33.9. Vemos também que, a média é de 20.09.
Vejamos agora um histograma da variável dependente.
df |>
ggplot(aes(x=mpg)) +
geom_histogram(binwidth = 2)
df |>
ggplot(aes(x=mpg)) +
geom_histogram(aes(y=after_stat(density)), binwidth = 2) +
geom_density()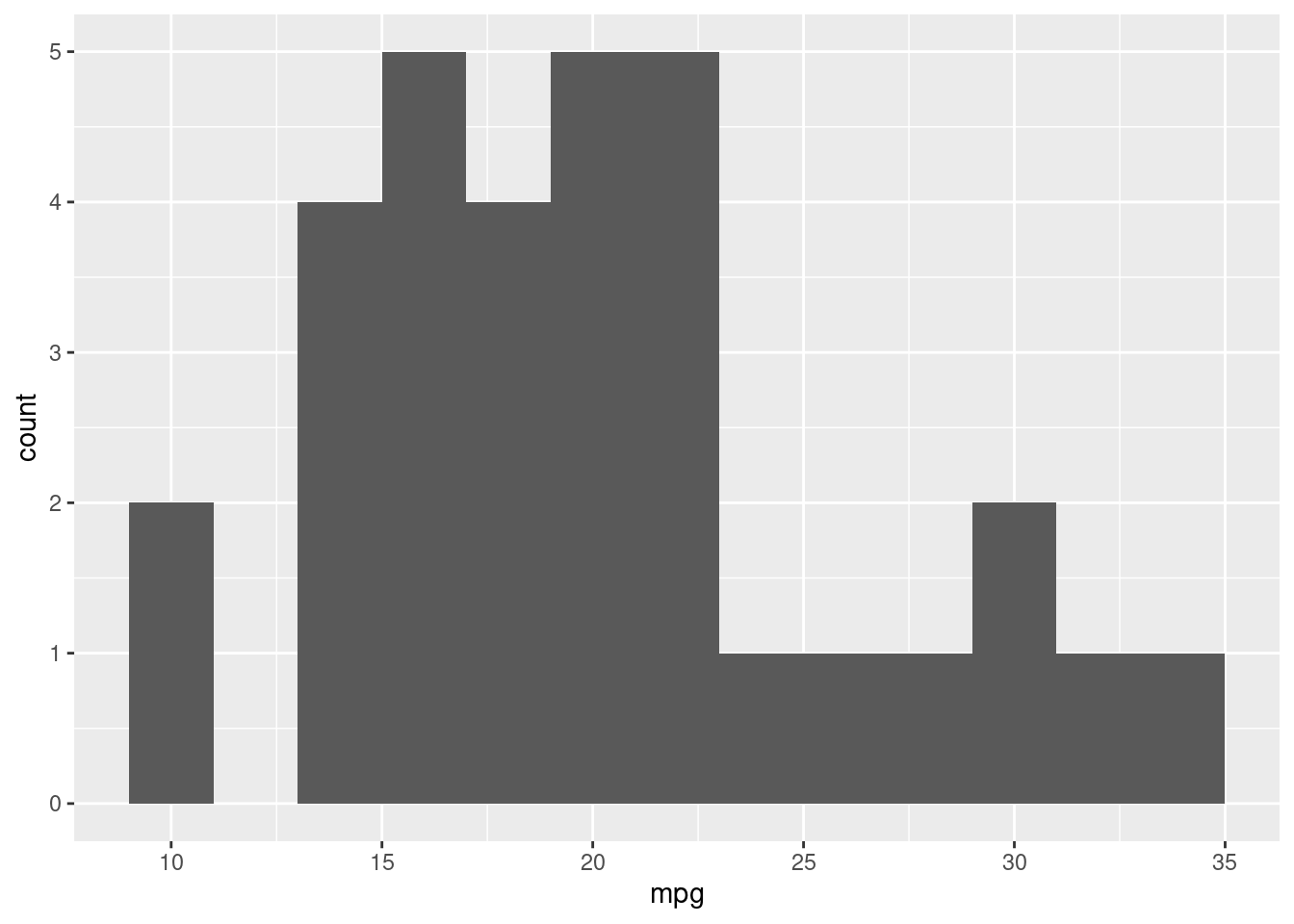
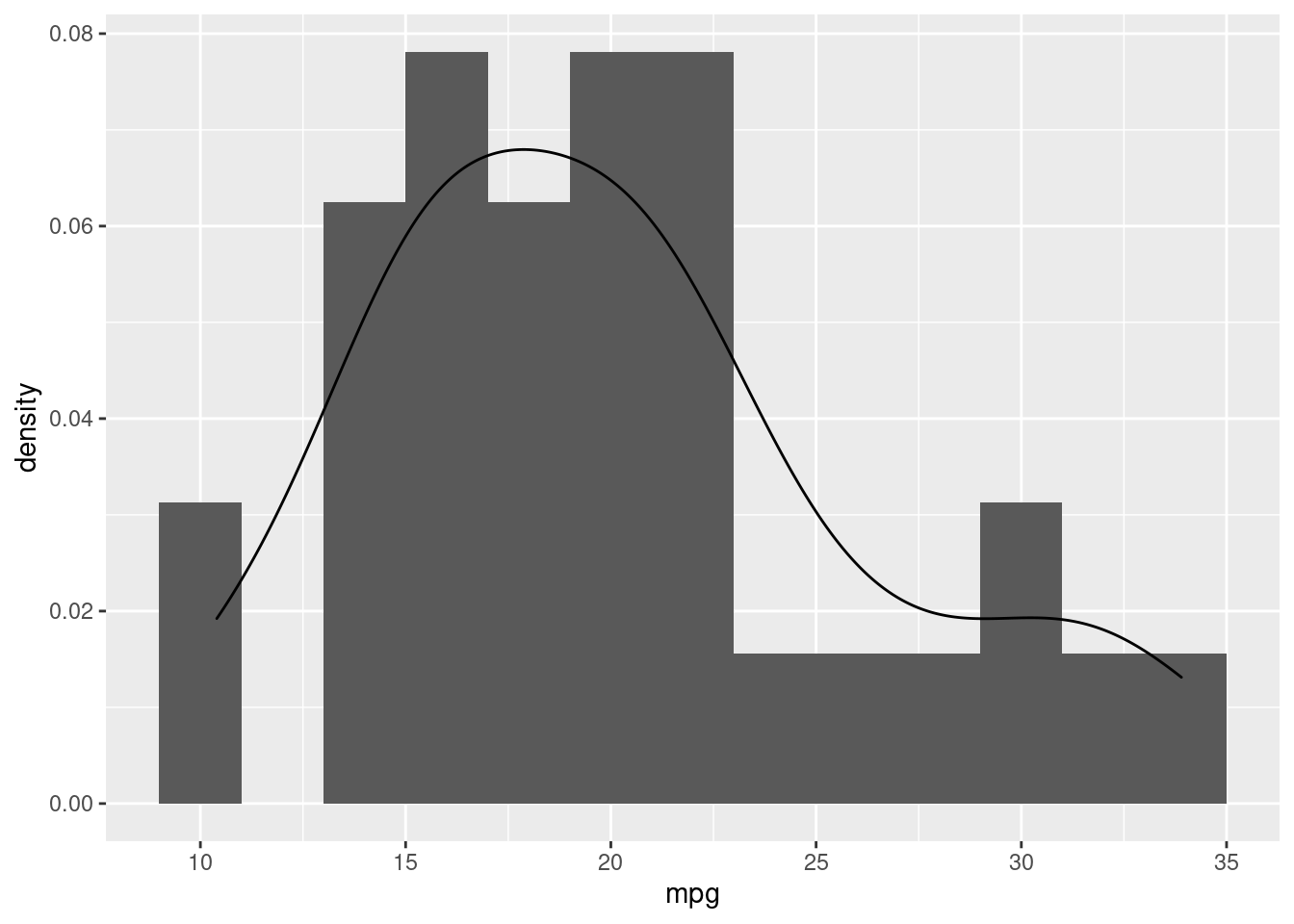
O gráfico da esquerda, nos mostra um histograma da variável dependente (mpg), ou seja, como as observações são distribuídas nas classes dentre os seus valores. Já o gráfico da direita, nos mostra também a estimativa de densidade kernel, que é uma forma não paramétrica de estimar a função de densidade de probabilidade (FDP) de uma vaiável. No eixo y, vemos valores que vão de 0 até 1, ou seja, de 0% até 100% de probabilidade.
Teste de Normalidade Shapiro-Francia
Podemos também, adicionar uma variável que segue a curva normal e adicioná-la ao gráfico anterior:
set.seed(123)
df$fp <- rnorm(rnorm(32), mean = mean(df$mpg), sd = sd(df$mpg))
df |>
ggplot(aes(x=mpg))+
geom_histogram(aes(y=after_stat(density)), binwidth = 2) +
geom_density() +
stat_function(aes(x=fp), fun = dnorm, args = list(mean = mean(df$fp), sd = sd(df$fp)),color = "blue")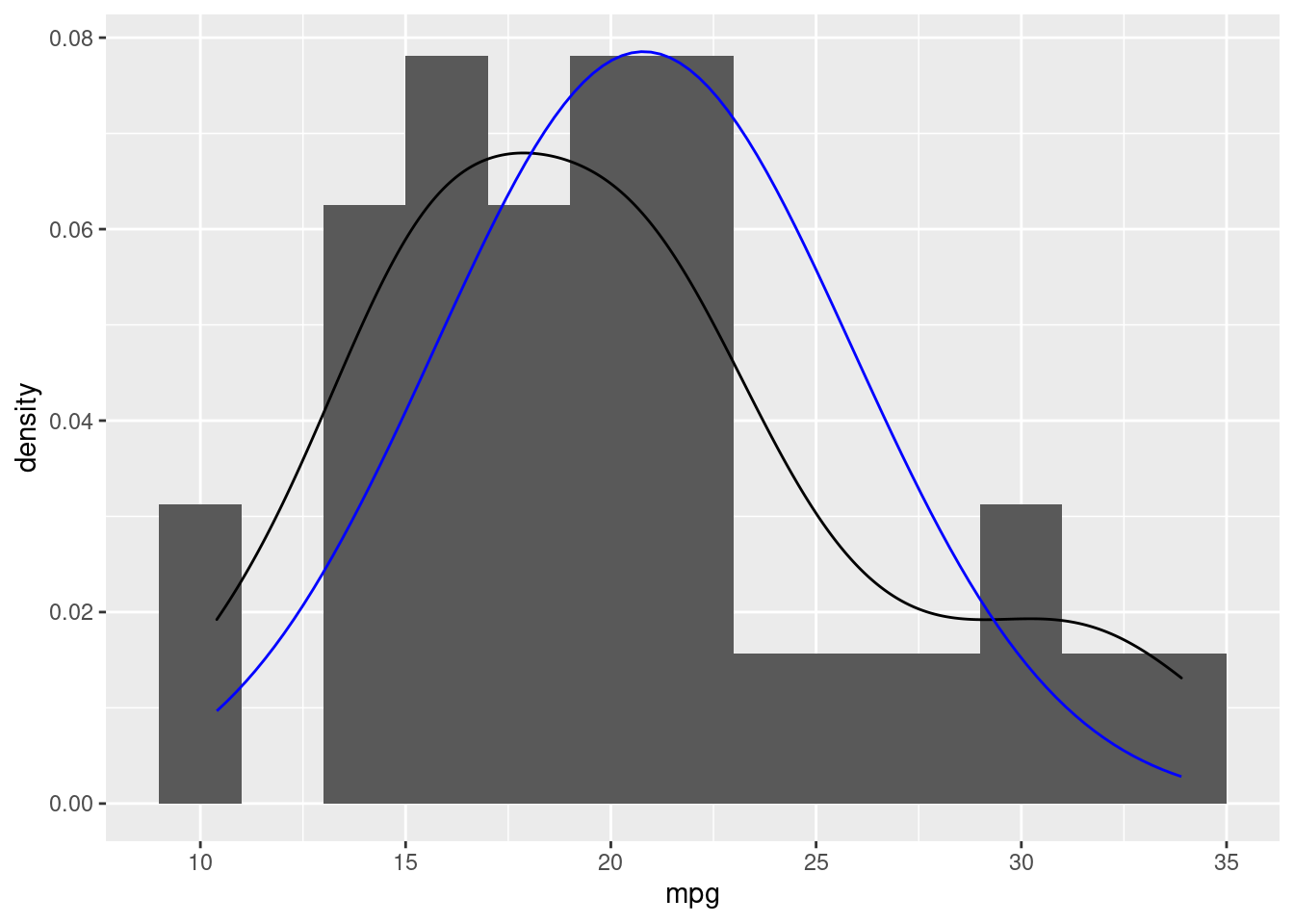
Aqui, observamos que nossa estimativa de densidade kernel (em preto) se aproxima de uma função distribuíção de probabilidade de uma curva normal (em azul). Apenas para sabermos se podemos afirmar que nossa variável dependente segue uma distribuíção normal, vamos fazer o teste estatístico de Shapiro-Francia.
Veja que esta NÃO é uma premissa para uso da técnica de regressão linear, isso é geralmente na validação dos resíduos conforme veremos mais adiante. Estamos apenas apresentando como fazer o teste e analisando a variável dependente.
# Teste de normalidade Shapiro-Francia
# p-valor <= 0.5 é não-normal, ou seja, maior a variável é normal
sf.test(df$mpg)
Shapiro-Francia normality test
data: df$mpg
W = 0.95247, p-value = 0.1495Neste caso, observamos que nossa variável dependente tem uma forma funcional aderente à uma normal.
Regressão Simples
Como já temos nossa variável dependente o consumo (mpg), iremos agora definir como variável explicativa, a potência (hp). A idéia é tentar criar um modelo preditivo em função apenas desta variável explicativa.
Correlação
Iremos inicialmente entender como a variável dependente (mpg) está relacionada com a variável explicativa (hp). Por se tratar de duas variáveis quantitativas, iremos utilizar a correlação de Pearson. Este é um coeficiente que informa o quão forte é esta correlação, variando de -1 até 1. Sendo: - Negativa (-1) - Quanto maior a variável dependente menor a variável explicativa - Neutra (0) - Não há correlação entre as variáveis - Positiva (1) - Quanto maior a variável dependente maior a variável
# Variável explicativa escolhida = hp
cor(df$mpg, df$hp)[1] -0.7761684DataExplorer::plot_correlation(df[c("mpg", "hp")])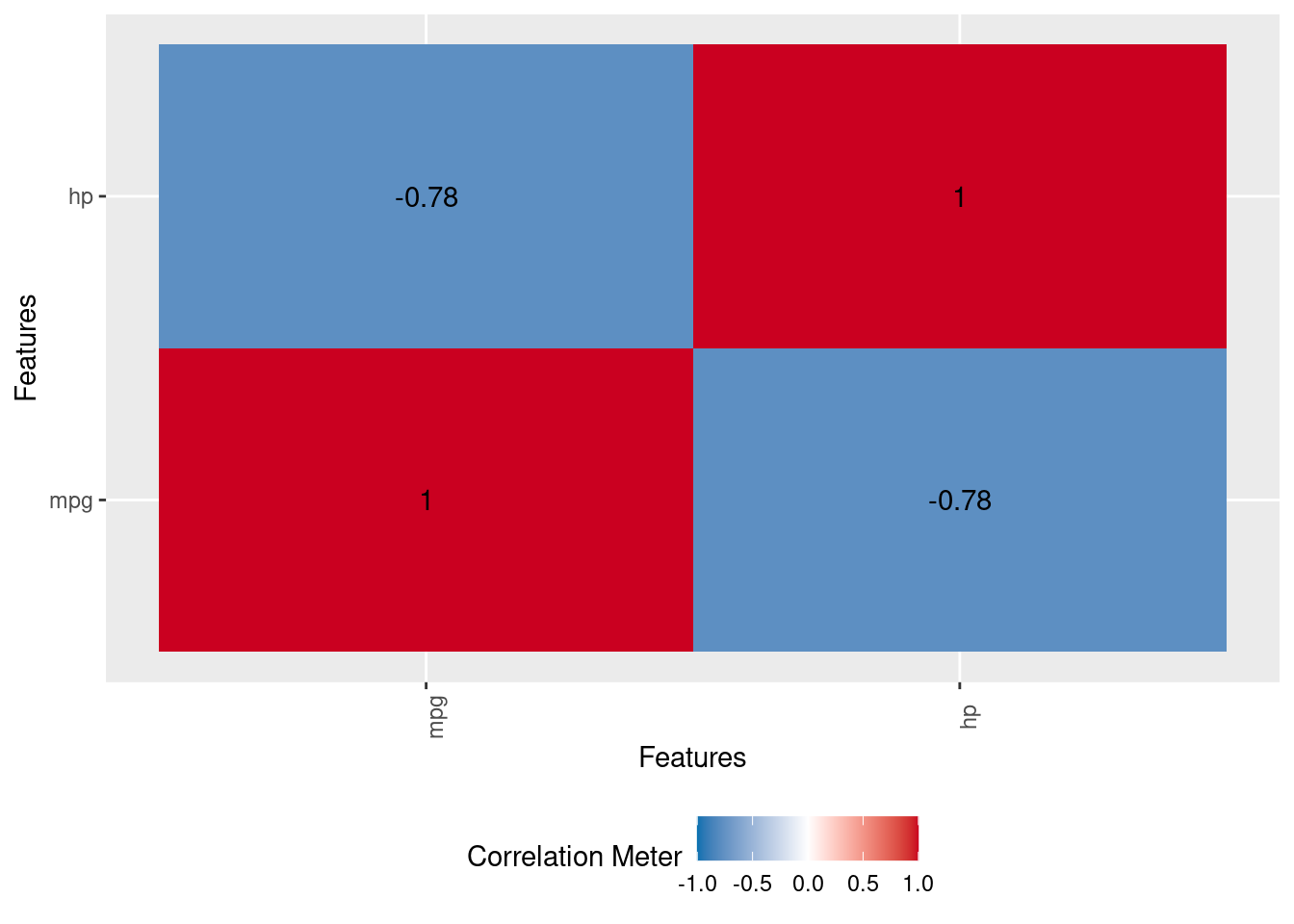
Nesta caso temos uma considerável correlação negativa (-0.78), ou seja, quanto maior a potência (hp), menor a quantidade de milhas por galão (mpg).
Gráfico de dispersão
Através do gráfico abaixo, podemos ter uma intuição visual do que representa a correlação de -0.78. Veja que se traçarmos uma reta atráves dos pontos, quanto maior o valor de mpg, menor o valor de hp e vice versa. Cada ponto no gráfico representa uma observação em nossa tabela de dados.
df |>
ggplot(aes(x = hp, y= mpg)) +
geom_point() +
geom_smooth(method = "lm", se = F)+
geom_text_repel(aes(label = name), size = 2, color = "darkgray")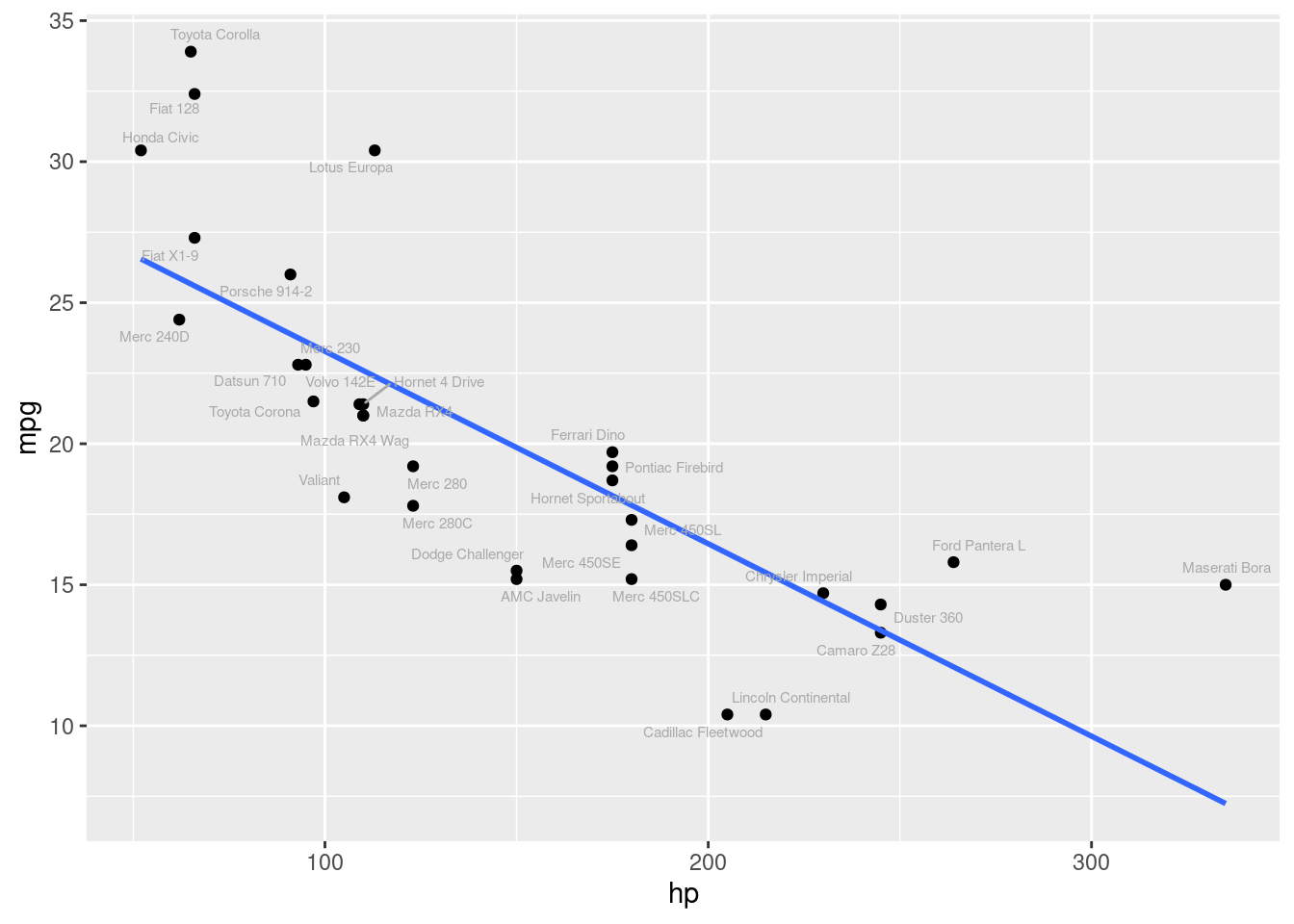
Criando um modelo linear simples
No caso de uma regressão linear simples, temos a seguinte equação que define a melhor reta entre os pontos do gráfico anterior. Entende-se como melhor reta aquela que apresenta a somatória dos resíduos igual à zero e a somatória dos resíduos ao quadrado for a mínima possível (Mean Square Error - MSE).
Função: \hat{Y}_i = \alpha + \beta * X1_i
O código abaixo, através da função “lm” irá criar um modelo de regressão linear simples, estimando os parâmetros para a equação de nossa reta. Neste caso, iremos utilizar o método “OLS” (Ordinary Least Square) para estimativa dos parâmetros desta equação. Não iremos cobrir os detalhes deste processo, mas há diversos métodos para encontrar estes coeficientes, como Mínimos Quadrados Ordinários (OLS), Máxima Verossimilhança (MLE), Descida do Gradiente, entre outros.
#Função lm para obter os coeficientes alpha e beta
modelo_uni <- lm(mpg ~ hp, data = df)
modelo_uni
Call:
lm(formula = mpg ~ hp, data = df)
Coefficients:
(Intercept) hp
30.09886 -0.06823 Onde, o \hat{Y} representa o valor previsto de nosso modelo, o \alpha o intercepto da reta, ou seja, que valor teórico teremos caso a variável explicativa fosse zero. Temos também o \beta que é inclinação da reta, ou seja, o quanto da variável explicativa é impactada em uma unidade. X1 neste caso, é o valor da nossa variável explicativa (hp).
Olhando estes coeficientes, podemos dizer que a cada 0.07 reduzida no valor da potência, aumentamos em uma milha por galão nosso consumo (mpg=miles per galon).
Neste caso, nossa função ficaria:
\hat{Y} = (30.09886) + [(-0.06823) * X1]
Ou seja, se quisermos prever o consumo (mpg) à partir apenas da variável explicativa potencia (hp), faríamos:
(30.09886) + [(-0.06823) * hp]
Por exemplo, de acordo com nosso modelo, para um veículo com 190 de potência, teremos:
(30.09886) + [(-0.06823) * 190] = (30.09886) - 12.9637 = \textbf{17.13516}
Ou seja, nosso modelo prevê um consumo de 17.13 milhas por galão se um veículo tiver 190 de potência.
Apenas como exemplo, vamos criar uma função que calcula a média das soma dos erros ao quadrado (Mean Square Error) e através da função optim() iremos buscar os valores de \alpha e \beta para que a soma dos erros ao quadrado seja menor possível.
funcao_perda <- function(X, y, par){
n <- length(y)
J <- sum((X%*%par - y)^2)/(2*n)
return (J)
}
X <- cbind(rep(1,32),df$hp)
y <- df$mpg
alpha_beta_iniciais <- c(0,0)
optim(par = alpha_beta_iniciais, X = X, y = y, fn = funcao_perda)$par
[1] 30.09641090 -0.06821044
$value
[1] 6.994912
$counts
function gradient
93 NA
$convergence
[1] 0
$message
NULLVeja que os coeficientes de \alpha = 30.09 e \beta = -0.068 são muito similares aos encontrados pela função lm().
Analizando o Modelo
Com o modelo criado anteriormente, podemos juntar nossas estimativas (aka fitted values) à base de dados originais e comparar nossos resultados:
media_mpg = round(summarise(df, m = mean(mpg))[[1]],1)
mpg_previsto = 17.13
hp_previsao = 190
bind_cols(df, modelo_uni$fitted.values) |>
rename (fitted = last_col()) |>
dplyr::select (orig = mpg, fitted, hp) |>
pivot_longer(cols = c("fitted", "orig")) |>
dplyr::rename (categoria = name, mpg = value) |>
ggplot(aes(x = hp, y = mpg, color = categoria)) +
geom_point(size = 3, alpha = 0.6) +
geom_point(x = hp_previsao, y= mpg_previsto, size = 5, color = "red")+
geom_vline(aes(xintercept = hp_previsao),
linetype = "dashed", color = "darkred")+
geom_text(aes(hp_previsao, mpg_previsto , label = paste("Previsto: ", mpg_previsto), vjust = -1, hjust = -0.5), color = "red")+
geom_hline(aes(yintercept = media_mpg),
linetype = "dashed", color = "navyblue")+
geom_text(aes(270,media_mpg, label = paste("Média: ", media_mpg), vjust = -1), show.legend = FALSE)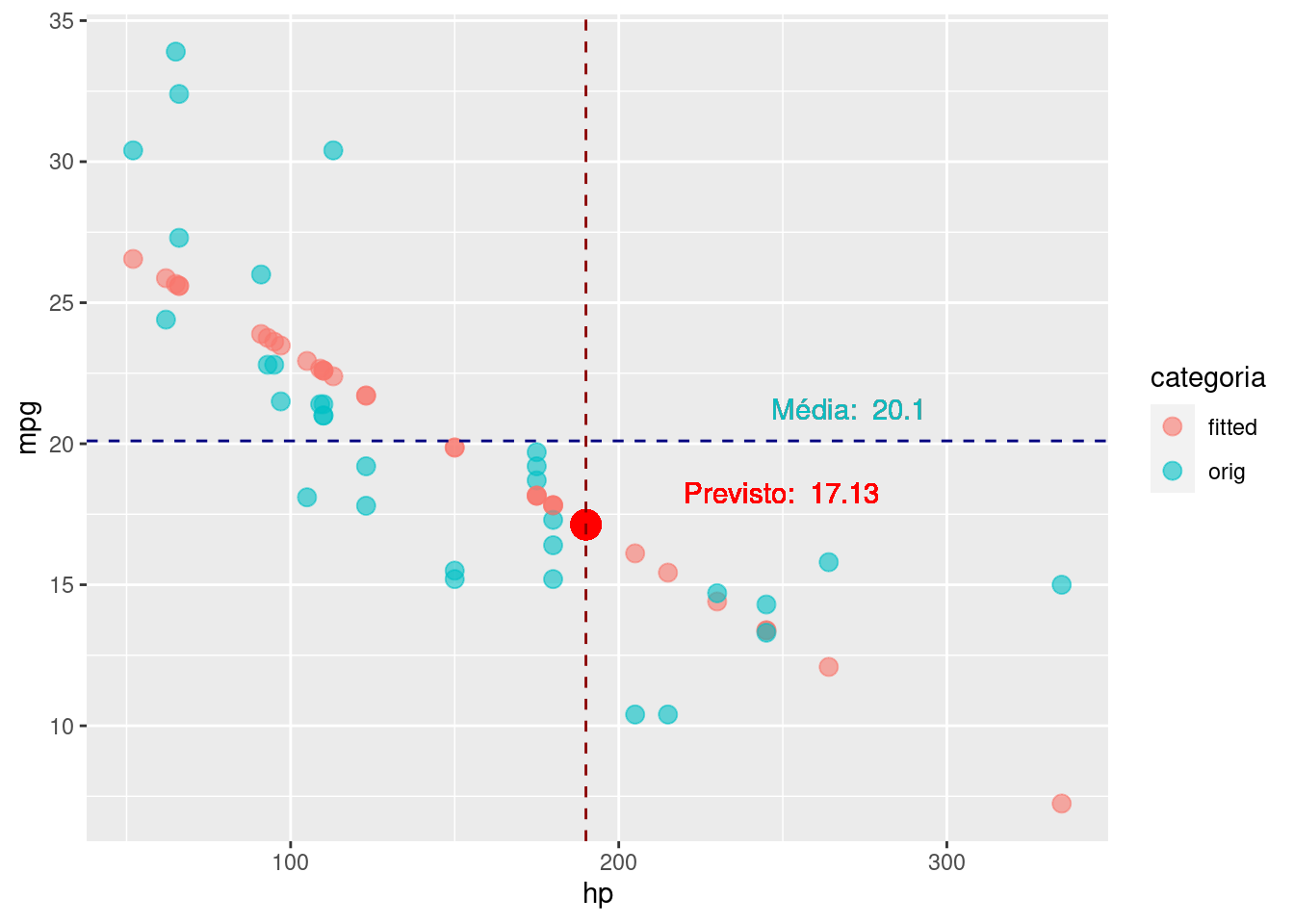
Veja que se tivéssemos escolhido apenas uma média da variável dependente (mpg), faríamos uma previsão de consumo de 20.1. Como utilizamos nosso modelo, nossa previsão mais acurada, prevendo um valor de 17.13.
Com o modelo criado anteriormente, podemos também utilizar a função summary() para extrair algumas informações bem importantes. Vejamos:
summary(modelo_uni)
Call:
lm(formula = mpg ~ hp, data = df)
Residuals:
Min 1Q Median 3Q Max
-5.7121 -2.1122 -0.8854 1.5819 8.2360
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 30.09886 1.63392 18.421 < 2e-16 ***
hp -0.06823 0.01012 -6.742 1.79e-07 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 3.863 on 30 degrees of freedom
Multiple R-squared: 0.6024, Adjusted R-squared: 0.5892
F-statistic: 45.46 on 1 and 30 DF, p-value: 1.788e-07Há diversos resultados a serem observados, dentre eles, podemos observar que a estatística F, com valor de p (p-value) menor que 0.05 basicamente nos indica que nosso modelo é estatisticamente melhor que um modelo onde não temos uma variável explicativa, ou seja, melhor que apenas usando a média dos valores conforme vimos anteriormente em nosso gráfico.
Observamos também que a cada -0.06823 de redução da potência (hp), temos uma economia de uma unidade de consumo (mpg).
A estatística T, também se mostra significante à 5% de significância para a variável (hp). Isto faz sentido, já que temos apenas esta variável em nosso modelo e já vimos que tínhamos modelo através da estatística F.
Usando a função predict().
Podemos utilizar a função predict para obter inferências do modelo criado ao invés do cálculo manual como fizemos anteriormente:
df_previsao = tibble("hp" = 190)
predict(modelo_uni, newdata = df_previsao) 1
17.13549 Visualizando a inferência feita pela função predict():
O gráfico abaixo, mostra nossa estimativa de consumo para um veículo de 190 hps como vimos anteriormente, porém agora com o resultado da função predict().
# Média da variável mpg:
df |> summarise(media = round(mean(mpg), 1))# Gráfico da estimativa do modelo e uma reta com a média da variável:
df |>
ggplot(aes(x = hp, y= mpg)) +
geom_point() +
geom_smooth(method = "lm", se = F)+
geom_abline(intercept = 20.1, slope = 0)+
geom_point(aes(x = 190, y = 20.1),color = "red", size = 3)+
geom_text_repel(aes(label = name), size = 2, color = "darkgray")+
geom_point(aes(x = 190, y = 17.13),color = "darkgreen", size = 3) 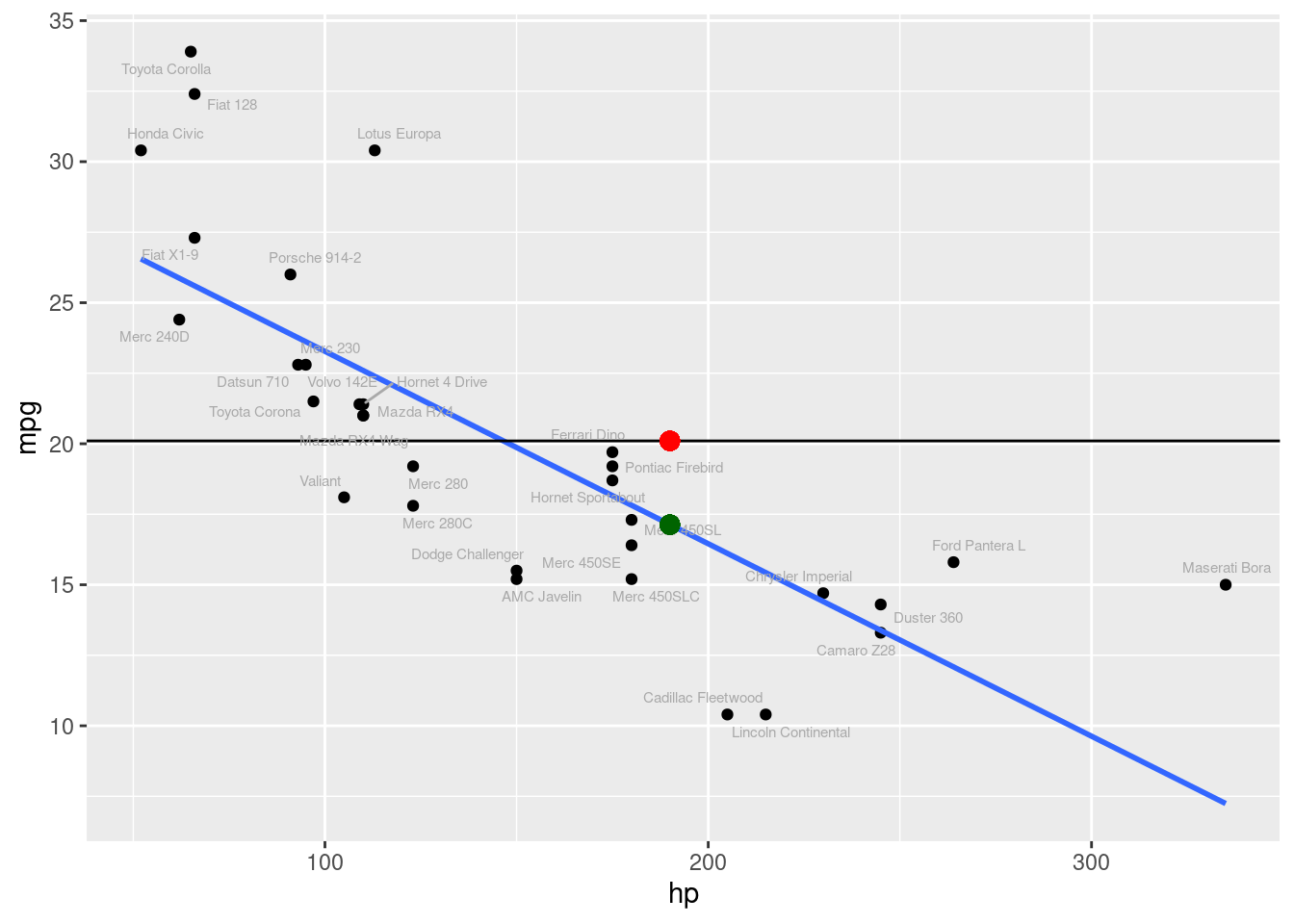
Coeficiente de ajuste do modelo R^2
O coeficiente R^2 é também chamado de coeficiente de ajuste do modelo, ou seja, o quanto da variância da variável dependente pode ser explicada pela variável explicativa.
Utilizamos a função summary() para obter seu valor. Também podemos utilizar a correlação de Pearson calculada anteriormente para calculá-lo, pois seu valor é a correlação de Pearson R ao quadrado.
#Obtendo o R2
summary(modelo_uni)$r.squared[1] 0.6024373#Validando o R2, extraindo a raiz, deve bater com a correlação anterior.
sqrt(summary(modelo_uni)$r.squared)[1] 0.7761684Variável Explicativa Qualitativa
Até o momento, criamos um modelo, onde a variável explicativa (X), era quantitativa. Mas e quando temos uma variável explicativa (X) qualitativa?
Digamos que iremos tentar prever nossa variável dependente (mpg) através da variável de números de cilindros (cyl). Se seguirmos os passos vistos até aqui, faríamos algo como, verificar sua correlação e depois criar um modelo de regressão simples. Vejamos o que poderia ocorrer:
Visualizando as correlações (ERRADO)
A seguir, iremos analisar as correlações e criar um modelo linear de forma similar à que fizemos até aqui.
CUIDADO!!! Estamos fazendo este procedimento de forma INCORRETA para mostrar alguns pontos importantes logo adiante.
df |> dplyr::select(mpg, cyl) |>
DataExplorer::plot_correlation()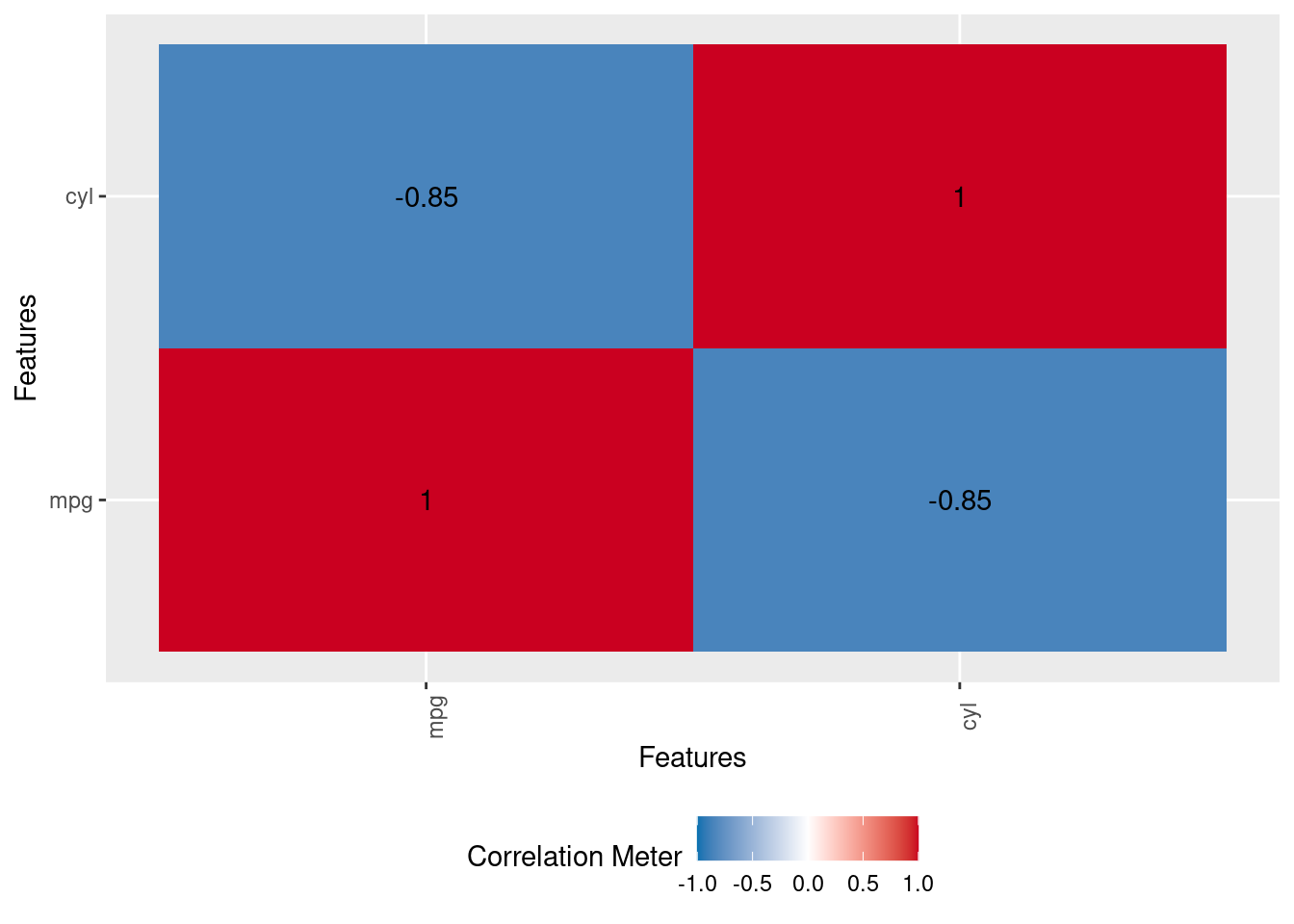
Olhando estas correlações, poderíamos entender que quanto menor o número de cilindros, mais econômico é o veículo. Esta afirmação não está necessariamente incorreta, mas vamos vizualizar as observações em termos de consumo (mpg) e número de cilindros (cyl) e depois criar um modelo.
df |>
ggplot(aes(x = cyl, y = mpg)) +
geom_point()+
geom_smooth(method = "lm", se=F)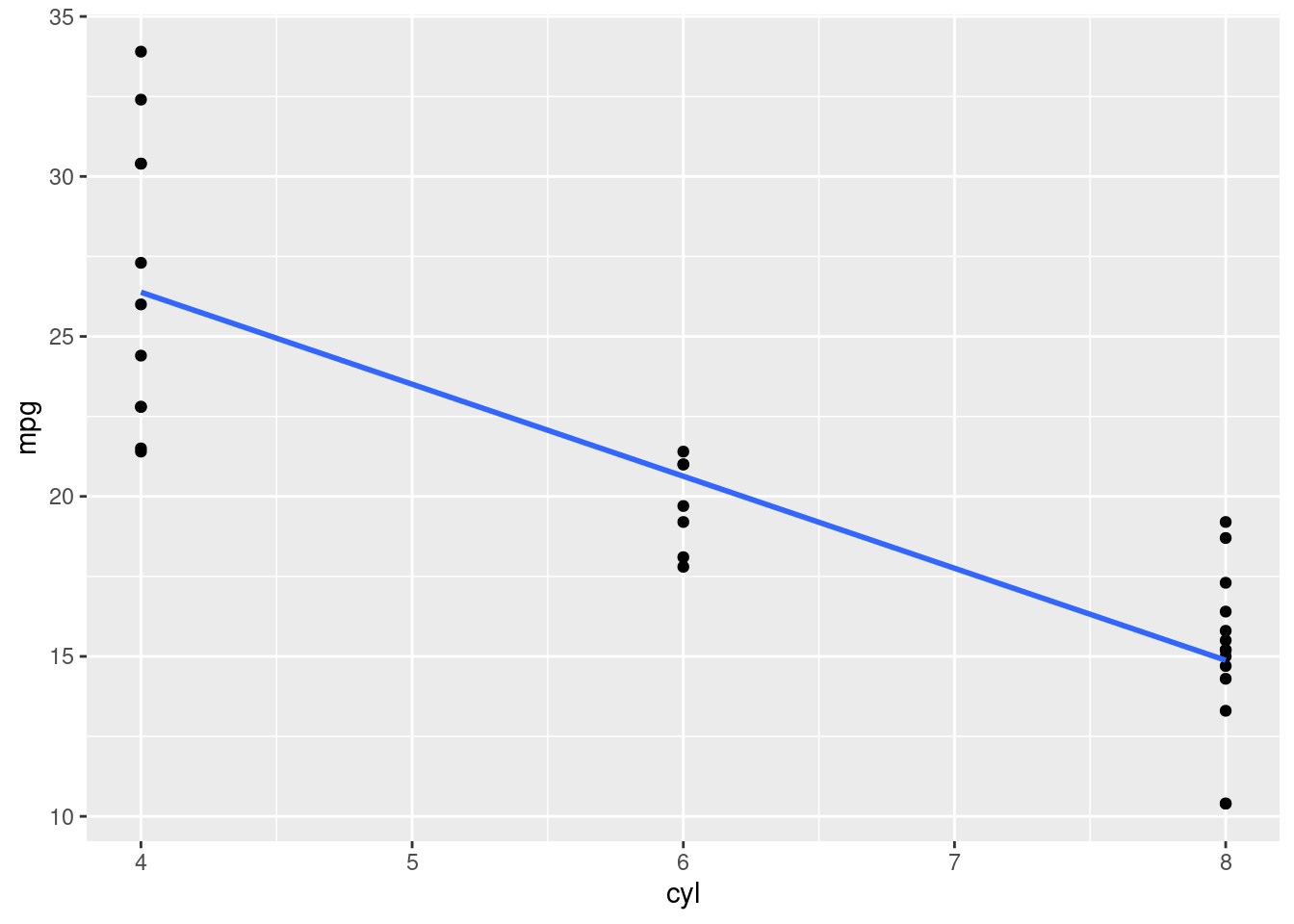
Criando o modelo (ERRADO)
Vamos criar um modelo linear simples, com o que vimos até aqui:
modelo_uni_errado <- lm(mpg ~ cyl, df)
summary (modelo_uni_errado)
Call:
lm(formula = mpg ~ cyl, data = df)
Residuals:
Min 1Q Median 3Q Max
-4.9814 -2.1185 0.2217 1.0717 7.5186
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 37.8846 2.0738 18.27 < 2e-16 ***
cyl -2.8758 0.3224 -8.92 6.11e-10 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 3.206 on 30 degrees of freedom
Multiple R-squared: 0.7262, Adjusted R-squared: 0.7171
F-statistic: 79.56 on 1 and 30 DF, p-value: 6.113e-10Vemos que os testes F e T tem seus p-valores menores que 5% (portanto passam nos testes de significância estatísticas). Vemos também que o R² nos diz que este modelo explica 73% da variância de nossa variável dependente.
Se seguirmos com a análise de nossos resultados do modelo, vemos que o valor do \beta está em -2.87. Isto, em tese, deveria nos dizer que, com uma redução de 2.87 cilindros, teríamos uma melhoria no consumo de uma unidade, ou seja, uma milha por galão (mpg). Oppssss…estranho, pois temos a possibilidade real de termos veículos de 4, 6 ou 8 cilindros.
Pode não ser tão evidente, mas o modelo criado é incorreto, pois a variável “cyl”, apesar de em nosso dataset estar configurada como “double” (quatitativa), ela é apenas uma categoria (“label”) para definir o tipo de cilindro é o automóvel, portanto é qualitativa.
Quando temos uma variável qualitativa, não temos média ou outras estatísticas de variáveis quantitativas. No máximo, podemos montar uma tabela frequência. Veja abaixo como ficaria a tabela de frequência (absoluta e relativa) da variável “cyl”
#Frequencia absoluta e relativa:
as_tibble(table(df$cyl),
.name_repair = "unique") |>
bind_cols(
enframe(prop.table(
table(df$cyl)))
) |> dplyr::select(num_cilindros = name, freq_absoluta = n, freq_relativa=value) |>
mutate (freq_relativa = scales::percent(as.numeric(freq_relativa)))Como nossa variável “cyl” na tabela, está como tipo double, a função lm(), está tratando seus valores numéricos, ou seja, as diferenças entre 4, 6 e 8 como se fosse uma variável quantitativa e isto está incorreto!!!
Ponderação Arbitrária
Aqui vale uma pequena pausa para entendermos melhor o que está acontecendo e seus impactos no modelo. Sabemos que devemos mudar a variável “cyl” que está originalmente quantitativa (4,6 e 8) para qualitativa. Porém, é um procedimento comum e incorreto atribuir valores de forma arbitrária, sendo estes, 1, 2 e 3 ou 4, 6 e 8, etc. Estes números são apenas “labels” para representar categorias desta variável.
Veja as médias adequadas quando mudamos a variável “cyl” como qualitativa:
df_cyl_medias <- df |> mutate (cyl = as_factor(cyl)) |>
group_by(cyl) |> summarise(mpg_media = mean(mpg))
df_cyl_mediasNeste caso, sabemos que em média, um veículo de 6 cilindros, tem um consumo de 19.7, enquanto que o de 4 e 8, tem respectivamente consumos médios de 26.7 e 15.1. Se tivessemos atribuído valores arbitrários, por exemplo, 4, 6 e 8, teríamos uma diferença de 2 entre cada um dos tipos de cilindros, o que é bem diferente do que vemos aqui.
Por exemplo: Veículos de 8 cilindros com diferença de 4.6 para os de 6 cilindros e 11.6 para os de 4 cilindros.
Visualizando as diferenças com ponderação arbitrária
Visualizando o modelo errado (com ponderação arbitrária de 1, 2 e 3 e as médias corretas (em vermelho):
df |>
ggplot(aes(x=cyl, y=mpg))+
geom_point()+
geom_text_repel(aes(label=name))+
geom_smooth(method="lm", se=F)+
geom_point(data = df_cyl_medias, aes(x=parse_number(levels(cyl)), y=mpg_media), color = "red")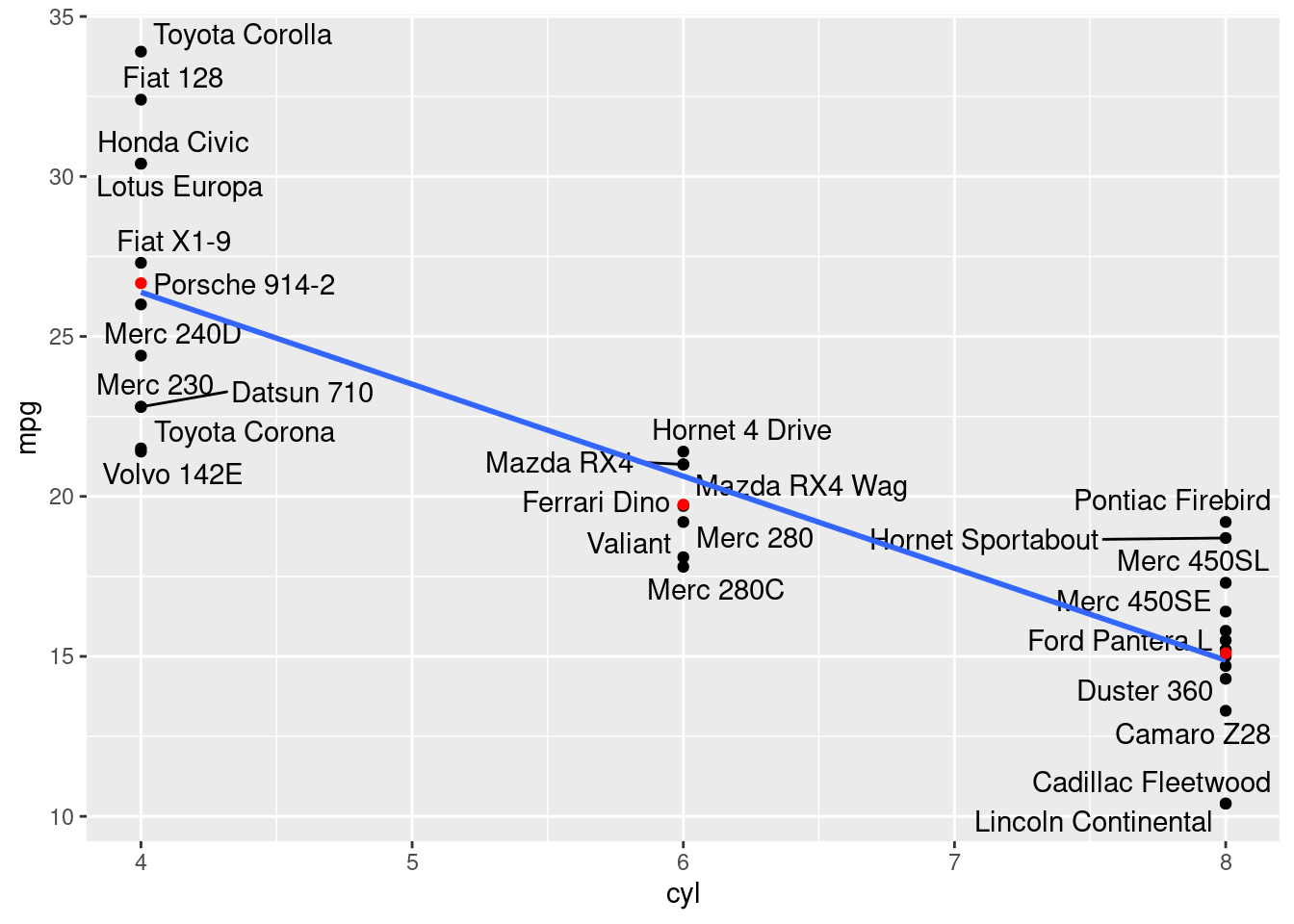
Observe como seria a inclinação dos betas considerando a frenquência média de cada categoria na variável cyl:
df |>
ggplot(aes(x=as_factor(cyl), y=mpg))+
geom_point()+
geom_text_repel(aes(label=name))+
geom_line(data = df_cyl_medias, aes(x=cyl, y=mpg_media,group =1), size =1.2,color = "blue")+
geom_point(data = df_cyl_medias, aes(x=cyl, y=mpg_media), color = "red")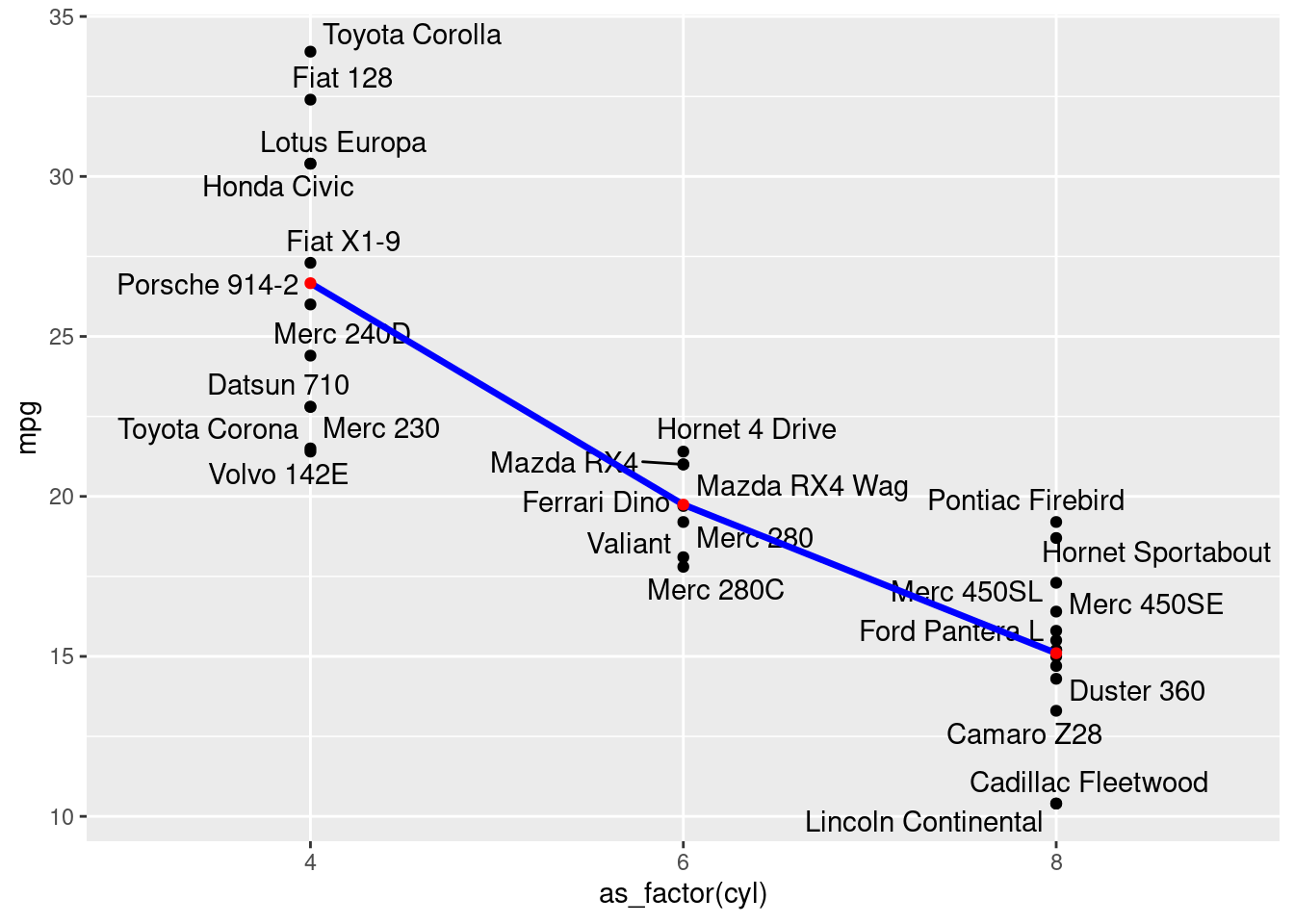
A discrepância entre os valores lineares trazedos por uma ponderação arbitrária e os respectivos valores quando utilizamos adequadamente a variável qualitativa pode trazer viéses muito maiores que neste simples exemplo, impactando de forma brutal a acurácia do modelo.
É por estre motivo que não podemos fazer a penderação arbitrária de valores para variáveis categóricas. Existe um procedimento adequado para lidar com esta situação que veremos a seguir.
Ajuste das variáveis qualitativas
Para adequar devidamente variáveis explicativas (x) categóricas para utilizarmos em modelos OLS, devemos criar variáveis adicionais “dummies”. Apesar da função lm() conseguir lidar com variáveis qualitativas ao utilizarmos fatores, faremos isto através da função dummy_columns() para detalhar o processo por trás deste método.
Iremos, a seguir, criar um data frame com a variável “cyl” como fator ao invés de double. Depois iremos utilzar a função dummy_collumns do pacote fastDummies para criar as variáveis dummies com base nos níveis dos fatores presentes:
df_fct <- df |> mutate (cyl = as_factor(cyl))
df_fct_dummy <- fastDummies::dummy_columns(df_fct, select_columns = "cyl",
remove_selected_columns = T,
remove_most_frequent_dummy = F,
remove_first_dummy = T)
glimpse(df_fct_dummy)Rows: 32
Columns: 14
$ name <chr> "Mazda RX4", "Mazda RX4 Wag", "Datsun 710", "Hornet 4 Drive", "H…
$ mpg <dbl> 21.0, 21.0, 22.8, 21.4, 18.7, 18.1, 14.3, 24.4, 22.8, 19.2, 17.8…
$ disp <dbl> 160.0, 160.0, 108.0, 258.0, 360.0, 225.0, 360.0, 146.7, 140.8, 1…
$ hp <dbl> 110, 110, 93, 110, 175, 105, 245, 62, 95, 123, 123, 180, 180, 18…
$ drat <dbl> 3.90, 3.90, 3.85, 3.08, 3.15, 2.76, 3.21, 3.69, 3.92, 3.92, 3.92…
$ wt <dbl> 2.620, 2.875, 2.320, 3.215, 3.440, 3.460, 3.570, 3.190, 3.150, 3…
$ qsec <dbl> 16.46, 17.02, 18.61, 19.44, 17.02, 20.22, 15.84, 20.00, 22.90, 1…
$ vs <dbl> 0, 0, 1, 1, 0, 1, 0, 1, 1, 1, 1, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 0…
$ am <dbl> 1, 1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 0, 0…
$ gear <dbl> 4, 4, 4, 3, 3, 3, 3, 4, 4, 4, 4, 3, 3, 3, 3, 3, 3, 4, 4, 4, 3, 3…
$ carb <dbl> 4, 4, 1, 1, 2, 1, 4, 2, 2, 4, 4, 3, 3, 3, 4, 4, 4, 1, 2, 1, 1, 2…
$ fp <dbl> 25.48550, 25.38309, 25.04225, 24.24102, 23.42906, 19.71749, 18.2…
$ cyl_6 <int> 1, 1, 0, 1, 0, 1, 0, 0, 0, 1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ cyl_8 <int> 0, 0, 0, 0, 1, 0, 1, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 1…Neste caso, ele atribui a existência (1) ou não existência (0) para cada categoria da variável quantitativa -1. No caso da variável cilindro (cyl), teríamos 3 variáveis, uma para cada categoria. Este procedimento é chamado de “one-hot-encoding”. Porém, como sempre uma variável é resultante da ausência das demais, o valor final, será sempre (1-n\;dummies). Em geral, utilizamos a categoria que possui a maior frequência (8 cilindros) como categoria de referência (parâmetro remove_most_frequent_dummy), apenas para exemplificar, escolhemos remover a primeira categoria (4 cilindros) com o parâmetro remove_first_dummy.
Agora temos 2 novas variáveis dummies (cyl_6 e cyl_8), sendo que a categoria de referência (cyl_4) é incluída no alpha da equação.
Neste caso, nossa equação ficaria:
\hat y_i = \alpha + \beta_1 *cyl\_6_i+\beta_2*cyl\_8_i
Com isso, sempre que tivermos zeros para cyl_6 e cyl_8, automaticamente sabemos que a categoria correponde a cyl_4.
Criando o modelo com dummy
Agora com a variável “cyl” devidamente “dummizada” podemos criar o modelo:
modelo_uni_dummy <- lm(mpg ~ cyl_6 + cyl_8, df_fct_dummy)
summary (modelo_uni_dummy)
Call:
lm(formula = mpg ~ cyl_6 + cyl_8, data = df_fct_dummy)
Residuals:
Min 1Q Median 3Q Max
-5.2636 -1.8357 0.0286 1.3893 7.2364
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 26.6636 0.9718 27.437 < 2e-16 ***
cyl_6 -6.9208 1.5583 -4.441 0.000119 ***
cyl_8 -11.5636 1.2986 -8.905 8.57e-10 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 3.223 on 29 degrees of freedom
Multiple R-squared: 0.7325, Adjusted R-squared: 0.714
F-statistic: 39.7 on 2 and 29 DF, p-value: 4.979e-09Apesar de termos feito o processo de dummies, saiba que a função lm() é inteligente o suficiente e já faz este processo quando recebe uma variável factor. Veja:
#A função lm() entende quando uma variável é factor como qualitativa e cria as dummies.
modelo_uni_fct <- lm(mpg ~ cyl, df_fct)
summary (modelo_uni_fct)
Call:
lm(formula = mpg ~ cyl, data = df_fct)
Residuals:
Min 1Q Median 3Q Max
-5.2636 -1.8357 0.0286 1.3893 7.2364
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 26.6636 0.9718 27.437 < 2e-16 ***
cyl6 -6.9208 1.5583 -4.441 0.000119 ***
cyl8 -11.5636 1.2986 -8.905 8.57e-10 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 3.223 on 29 degrees of freedom
Multiple R-squared: 0.7325, Adjusted R-squared: 0.714
F-statistic: 39.7 on 2 and 29 DF, p-value: 4.979e-09Neste caso, vemos que a estatística F passa no teste à 5% de significância, ou seja, temos um modelo. Vemos também que às dummies para 6 e 8 cilindros também passam no teste.
Ao ler os coeficientes, devemos sempre comparar com a categoria de referência. Por exemplo, podemos dizer que automóveis com 8 cilindros consomem 11.56 milhas por galão que automóveis de 4 cilindros, considerando todas as demais variáveis idênticas.
prev <- tribble(~cyl,
factor(4, levels = c(4,6,8)),
factor(6, levels = c(4,6,8)),
factor(8, levels = c(4,6,8)))
predict(modelo_uni_fct, prev) 1 2 3
26.66364 19.74286 15.10000 Regressão Multivariada
Até aqui, sempre criamos um modelo de predição com base em apenas uma variável explicativa. Veremos como podemos adicionar mais variáveis explicativas para aumentar a acurácia de nosso modelo. Para exemplificar iremos adicionar a variável explicativa peso (wt).
Correlações
Por se tratar de outra variável quantitativa, iremos utilizar a correlação de Pearson para entender como estas variáveis se correlacionam:
#Desta vez, iremos utilzar a função Chart.correlation do pacote PerformanceAnalytics
PerformanceAnalytics::chart.Correlation(df[c("mpg", "hp", "wt")])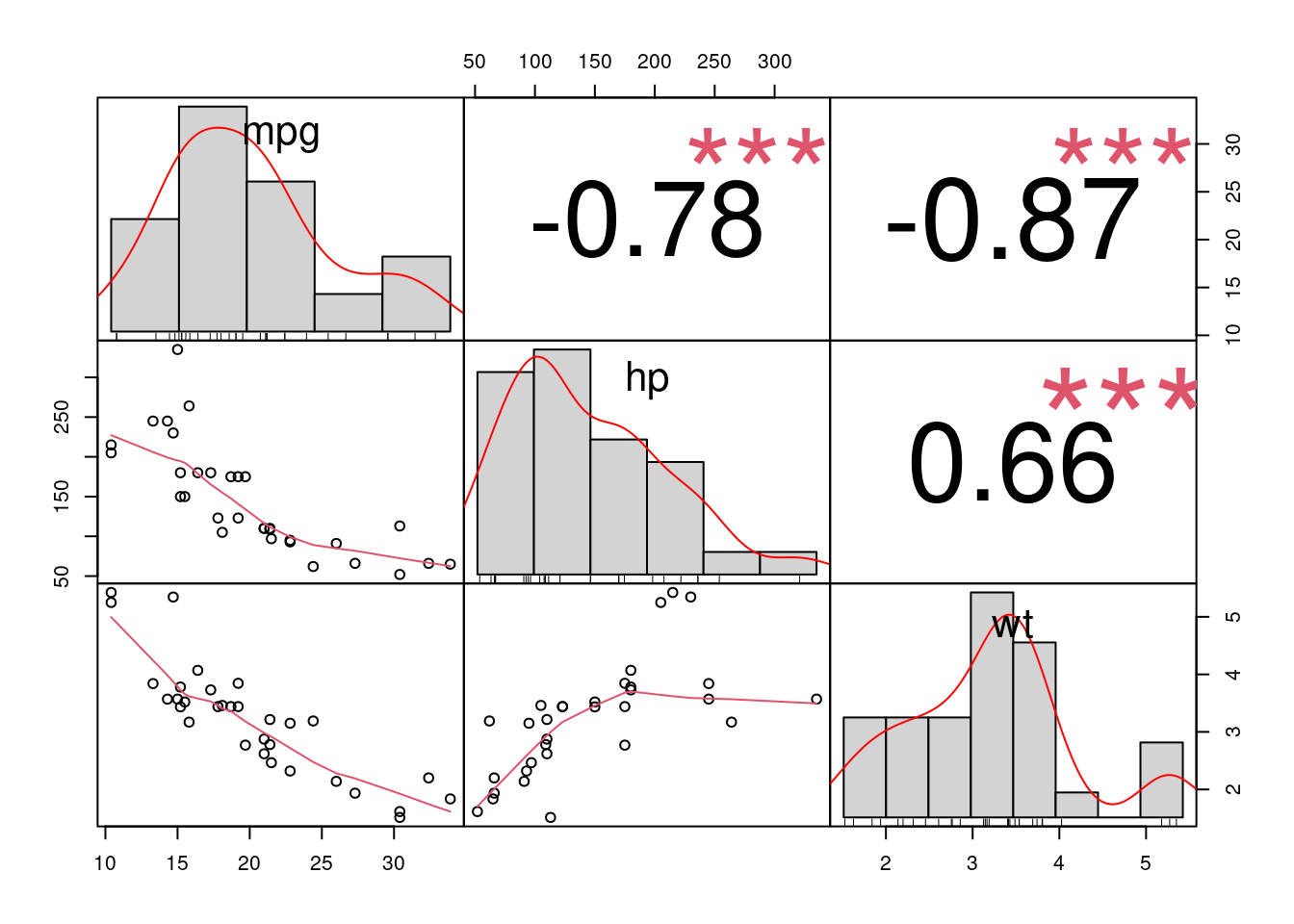
Assim como a potência (hp), em nosso exemplo inicial, vemos que há uma considerável correlação negativa com a variável peso (wt) de -0.87.
Da mesma maneira que a regressão linear simples, também temos a equação que define uma regressão linear multivariada:
Função: \hat{Y}_i = \alpha + \beta_1 * X_{1i} + \beta_2 * X_{2i} + ...+\beta_{K} * X_{K_i}
Criando um modelo multivariado
Iremos agora utilizar nossa função lm() para criar o modelo multivariado.
#Função lm para obter os coeficientes alpha e beta das duas variáveis (hp e wt)
modelo_multi_1<- lm(mpg ~ hp + wt, data = df)
modelo_multi_1
Call:
lm(formula = mpg ~ hp + wt, data = df)
Coefficients:
(Intercept) hp wt
37.22727 -0.03177 -3.87783 Através da função summary() podemos observar que tanto a estatística F (do modelo), quanto as duas variáveis (estatística T) passam com um grau de confiança de 95%:
summary(modelo_multi_1)
Call:
lm(formula = mpg ~ hp + wt, data = df)
Residuals:
Min 1Q Median 3Q Max
-3.941 -1.600 -0.182 1.050 5.854
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 37.22727 1.59879 23.285 < 2e-16 ***
hp -0.03177 0.00903 -3.519 0.00145 **
wt -3.87783 0.63273 -6.129 1.12e-06 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 2.593 on 29 degrees of freedom
Multiple R-squared: 0.8268, Adjusted R-squared: 0.8148
F-statistic: 69.21 on 2 and 29 DF, p-value: 9.109e-12Comparando os modelos
Podemos utilizar o R^2 ajustado para comparar o coeficiente de ajuste dos modelos:
summary(modelo_uni)$adj.r.squared[1] 0.5891853summary(modelo_multi_1)$adj.r.squared[1] 0.8148396Neste caso, observamos que adicionando a variável peso, conseguimos explicar 81% da variância da variável dependente de consumo (mpg) ao invés dos 59% quando tínhamos apenas a variável pontência (hp).
Resíduos e Homocedasticidade
A regressão linear possui algumas premissas que devem ser observadas. Os gráficos a seguir, nos ajudam a entender se os resíduos seguem uma distribuição normal, se há outliers, e se temos homocedasticidade, ou seja, variância homogênia nos resíduos. Estas premissas são importantes para a acurácia de uma modelo de regressão linear.
par(mfrow=c(2,2))
plot (modelo_multi_1)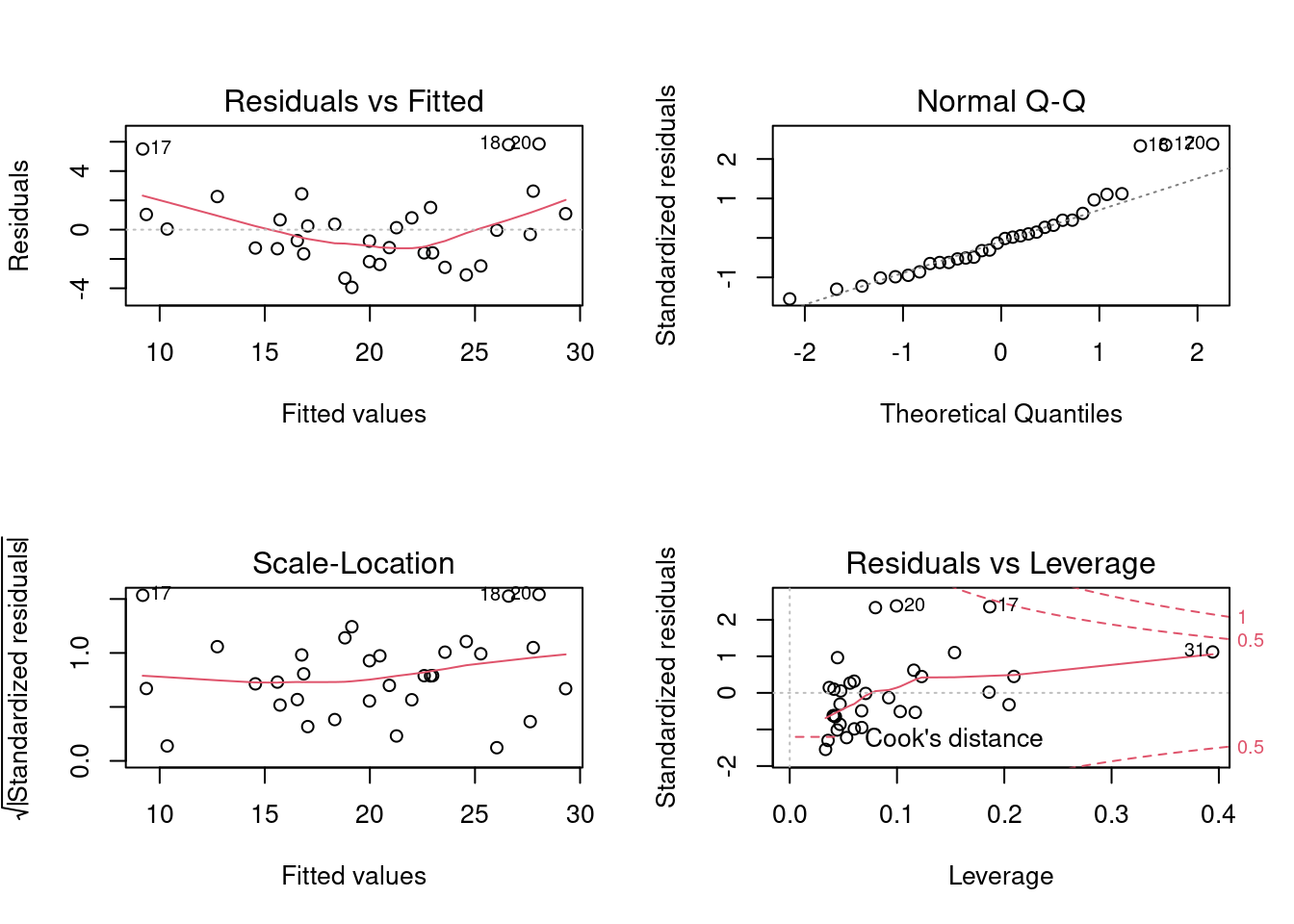
Para validar a leitura dos gráficos anteriores, podemos utilizar os testes estatísticos a seguir:
Normalidade dos resíduos
# Teste sharpiro-francia (ou sharpiro-wilk para amostrar < 30)
shapiro.test(modelo_multi_1$residuals)
Shapiro-Wilk normality test
data: modelo_multi_1$residuals
W = 0.92792, p-value = 0.03427Como podemos observar, nossos resíduos não passam no teste de normalidade (p-value <= 0.05). A seguir deixaremos os demais testes com o código, mas devemos fazer algo a respeito desta premissa não atendida.
Outliers nos resíduos:
# Os resíduos PADRONIZADOS devem estar entre -3 e +3 e mediana perto de zero
summary(rstandard(modelo_multi_1)) Min. 1st Qu. Median Mean 3rd Qu. Max.
-1.54564 -0.63032 -0.07358 0.01855 0.44828 2.37862 No caso acima, não temos outliers nos resíduos
# Independência dos resíduos. Não se aplicaria aqui, mas deixamos o código. Em geral, quando temos análise longitudinal (medidas repetidas), ex. Time series.
# Recomendação de 1 e 3. p-value > 0.05 os resíduos são independentes. Teste adequando quando os resíduos atendem a normalidade.
car::durbinWatsonTest(modelo_multi_1) lag Autocorrelation D-W Statistic p-value
1 0.2954091 1.362399 0.05
Alternative hypothesis: rho != 0Homocedasticidade:
#Teste de Breush-Pagan. Também tem premissa de normalidade nos resíduos.
#H0 existe homocedasticidade e H1 Não existe, ou sejá, tem Heterocedasticidade.
# Neste caso, p-valor > 0.05, portanto temos homocedasticidade.
lmtest::bptest(modelo_multi_1)
studentized Breusch-Pagan test
data: modelo_multi_1
BP = 0.88072, df = 2, p-value = 0.6438Como nossa premissa de normalidade dos resíduos não foi antendida, podemos tentar fazer um transformação não linear em nossa variável dependente.
Transformação de Box-Cox
Ao fazer uma transformação na variável dependente através de uma Transformação de Box-Cox, podemos ter uma variação mais uniforme. Vamos testar, criando um modelo atraveś de uma nova variável que possui uma transformação de Box-Cox da variável mpg e iremos analizar seus resíduos.
df2 <- df
#Transformação de Box-Cox
#Estimando o lambda de BoxCox
lambda_BC <- car::powerTransform(df2$mpg)
lambda_BC Estimated transformation parameter
df2$mpg
0.02956537 #Adicionando na base de dados: -->
df2$mpg_bc <- (((df2$mpg ^ lambda_BC$lambda) - 1) / lambda_BC$lambda)
modelo_multi_2_bc <- lm(mpg_bc~ hp + wt, df2)
summary(modelo_multi_2_bc)
Call:
lm(formula = mpg_bc ~ hp + wt, data = df2)
Residuals:
Min 1Q Median 3Q Max
-0.20511 -0.08248 -0.02739 0.06790 0.31116
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 4.0428360 0.0750633 53.859 < 2e-16 ***
hp -0.0016878 0.0004239 -3.981 0.000421 ***
wt -0.2185744 0.0297069 -7.358 4.18e-08 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.1218 on 29 degrees of freedom
Multiple R-squared: 0.8687, Adjusted R-squared: 0.8596
F-statistic: 95.91 on 2 and 29 DF, p-value: 1.646e-13Agora vamos visualizar e testar a normalidade dos resíduos:
par(mfrow=c(2,2))
plot (modelo_multi_2_bc)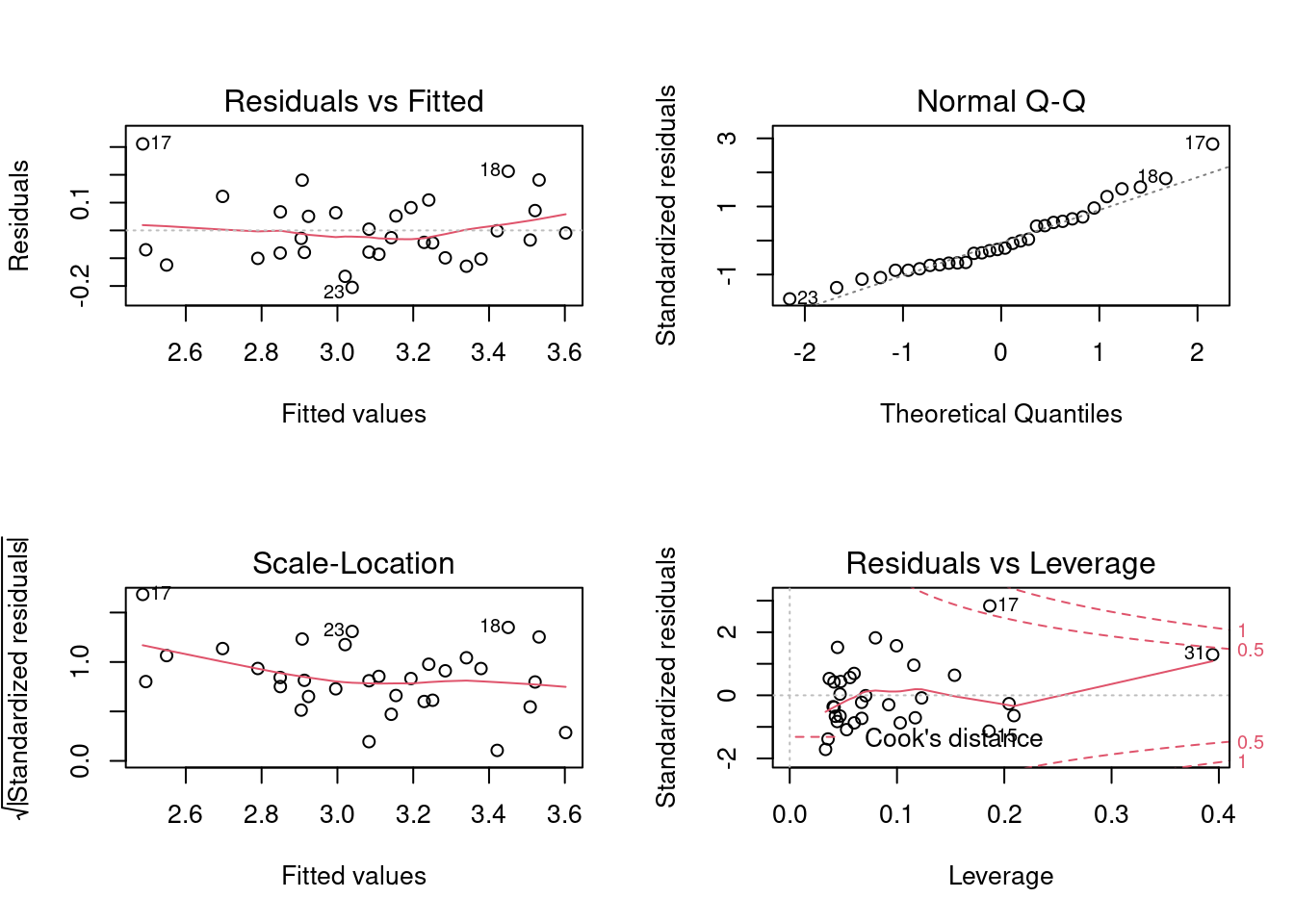
Testes de Normalidade, Homocedasticidade e Outliers:
# Teste sharpiro-francia (ou sharpiro-wilk para amostrar < 30)
sf.test(modelo_multi_2_bc$residuals)
Shapiro-Francia normality test
data: modelo_multi_2_bc$residuals
W = 0.95845, p-value = 0.2145Como podemos observar, nossos resíduos agora passam no teste de normalidade (p-value > 0.05). .
Outliers nos resíduos:
# Os resíduos PADRONIZADOS devem estar entre -3 e +3 e mediana perto de zero
summary(rstandard(modelo_multi_2_bc)) Min. 1st Qu. Median Mean 3rd Qu. Max.
-1.71335 -0.71578 -0.24297 0.01259 0.58248 2.83329 No caso acima, não temos outliers nos resíduos
# Independência dos resíduos. Não se aplicaria aqui, mas deixamos o código. Em geral, quando temos análise longitudinal (medidas repetidas), ex. Time series.
# Recomendação de 1 e 3. p-value > 0.05 os resíduos são independentes. Teste adequando quando os resíduos atendem a normalidade.
car::durbinWatsonTest(modelo_multi_2_bc) lag Autocorrelation D-W Statistic p-value
1 0.1855612 1.601514 0.2
Alternative hypothesis: rho != 0Homocedasticidade:
#Teste de Breush-Pagan. Também tem premissa de normalidade nos resíduos.
#H0 existe homocedasticidade e H1 Não existe, ou sejá, tem Heterocedasticidade.
# Neste caso, p-valor > 0.05, portanto temos homocedasticidade.
lmtest::bptest(modelo_multi_2_bc)
studentized Breusch-Pagan test
data: modelo_multi_2_bc
BP = 3.2947, df = 2, p-value = 0.1926Salvando os fitted values Box-Cox:
Como fizemos o modelo com os valores transformados, precisamos lembrar de aplicar a fórmula inversa para termos o valor de mpg adequado:
df2$mpg_bc_fitted <- (((modelo_multi_2_bc$fitted.values*(lambda_BC$lambda))+
1))^(1/(lambda_BC$lambda))Vamos utilizar agora a função predict() para estimar nosso consumo com o novo modelo multivariado com a a transformação de Box-Cox para um veículo com 3000 libras de peso e 190 de potência:
df_previsao_bc = tibble("hp" = 190, "wt" = 3.0)
predict(modelo_multi_2_bc, newdata = df_previsao_bc) 1
3.06644 Veja que esta previsão é o valor transformado, portanto, para saber o valor correto, devemos fazer a operação inversa da transformação, ou seja, multiplicar pelo lambda e elevar a 1/lambda.
((3.06644 * (lambda_BC$lambda)) + 1)^(1/(lambda_BC$lambda)) df2$mpg
18.82727 Colocando direto no código, temos:
df_previsao_bc = tibble("hp" = 190, "wt" = 3.0)
previsao <- predict(modelo_multi_2_bc, newdata = df_previsao_bc)
((previsao * (lambda_BC$lambda)) + 1)^(1/(lambda_BC$lambda)) 1
18.82726 Visualizando a inferência
Neste caso, como temos duas variáveis explicativas, iremos criar um gráfico 3D para visualizar o hiper-plano da regressão:
plot_ly(df2, x= ~mpg, y=~hp, z=~wt) |>
add_markers(name = "Dados Treino") |>
add_markers(x = 18.83, y = 190, z = 3.0,
name = "Previsao") Multi-colinearidade e Stepwise
Temos um importante ponto a ser discutido quando se trata de modelos multi-variados. Se trata da multi-colinearidade e também das significâncias das variáveis na presença de outras variáveis explicativas.
Como vimos anteriormente, nosso modelo multivariado, utilizando as variáveis hp e wt, teve um melhor ajuste que nosso modelo simples utilzando apenas a variável hp. Digamos que agora, desejamos introduzir uma nova variável por acreditarmos que ela pode melhorar ainda mais nosso modelo. Ao olharmos as correlações com a variável dependente “mpg”, observamos que a variável deslocamento “disp” tem forte correlação negativa com a variável dependente.
cor(df$mpg, df$disp)[1] -0.8475514Com isso, criamos um modelo simples, apenas com esta variável, e vemos que ela não é somente significativa, mas também explica 72% da variância de mpg (R2 = .72).
summary(lm(mpg ~ disp, df))
Call:
lm(formula = mpg ~ disp, data = df)
Residuals:
Min 1Q Median 3Q Max
-4.8922 -2.2022 -0.9631 1.6272 7.2305
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 29.599855 1.229720 24.070 < 2e-16 ***
disp -0.041215 0.004712 -8.747 9.38e-10 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 3.251 on 30 degrees of freedom
Multiple R-squared: 0.7183, Adjusted R-squared: 0.709
F-statistic: 76.51 on 1 and 30 DF, p-value: 9.38e-10Com isso, decidimos introduzi-la em nosso modelo multivariado, juntamente com as variáveis hp e wt com a intenção de deixá-lo ainda melhor.
modelo_multi_3_autocor <- lm(mpg ~hp+wt+disp, df)
summary(modelo_multi_3_autocor)
Call:
lm(formula = mpg ~ hp + wt + disp, data = df)
Residuals:
Min 1Q Median 3Q Max
-3.891 -1.640 -0.172 1.061 5.861
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 37.105505 2.110815 17.579 < 2e-16 ***
hp -0.031157 0.011436 -2.724 0.01097 *
wt -3.800891 1.066191 -3.565 0.00133 **
disp -0.000937 0.010350 -0.091 0.92851
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 2.639 on 28 degrees of freedom
Multiple R-squared: 0.8268, Adjusted R-squared: 0.8083
F-statistic: 44.57 on 3 and 28 DF, p-value: 8.65e-11Depois de analisar seu resultado através da função summary(), vemos que ela não passa em nosso teste T, com p-value de 0.92851.
Isto ocorre, pois há uma forte correlação entre variáveis explicativas, neste caso entre disp e wt e um pouco menos forte entre disp e hp. Veja:
df |> dplyr::select (mpg, hp, wt, disp) |> correlation::correlation(method = "pearson") |> plot()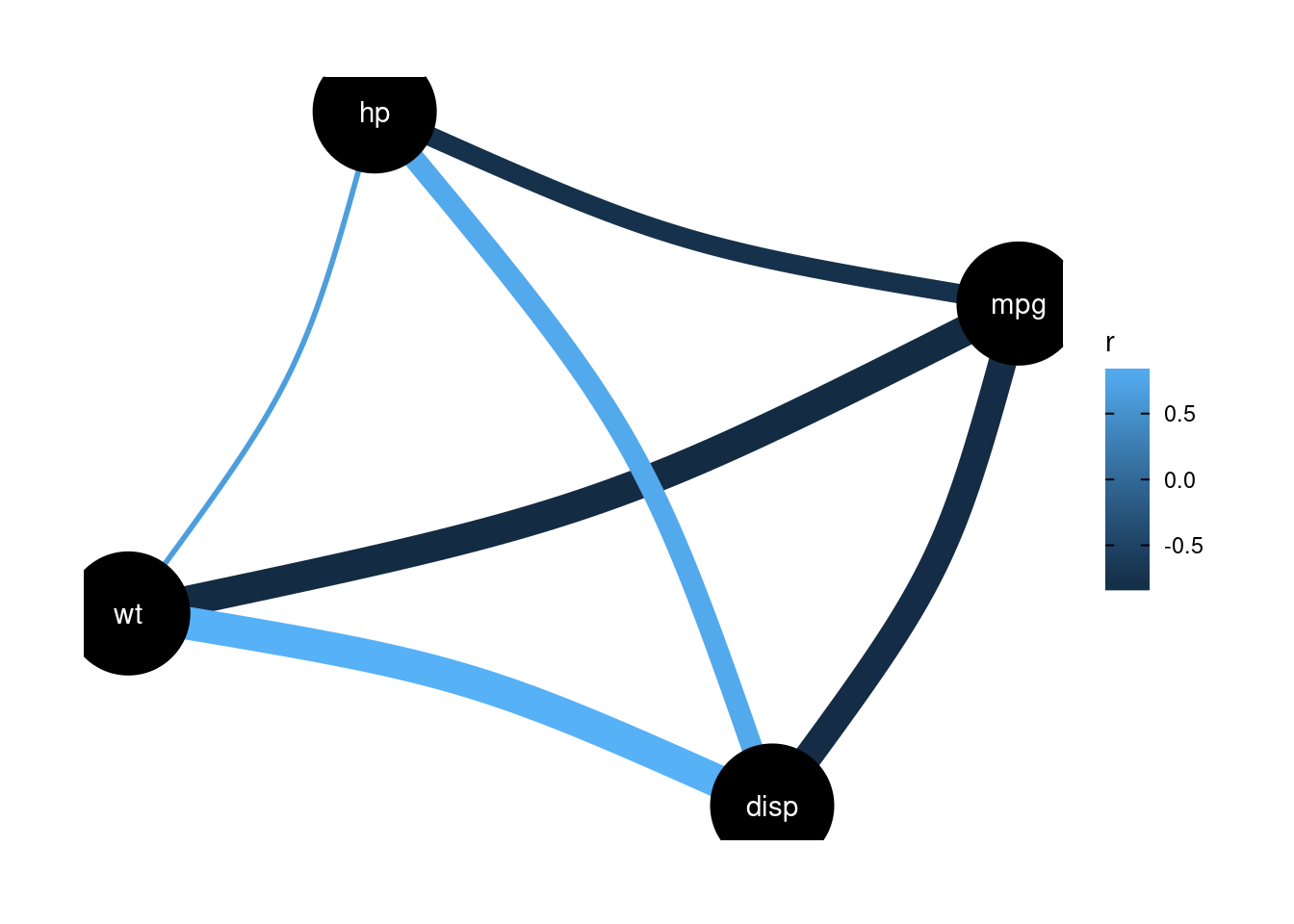
Devido à isto, a variável disp, se torna menos relevante na presença das demais.
A função ols_vif_tol() do pacote olsrr nos ajuda a fazer este disgnóstico de multi-colinearidade através do fator de Inflação de Variância (VIF) que vai de 1 até +(infinito) ou Tolerância (Tolerance) que vai de 0 até 1:
# Tol: quanto mais próximo de 1 melhor.
# VIF: quanto maior pior.
olsrr::ols_vif_tol(modelo_multi_3_autocor)Em modelos com muitas variáveis explicativas, pode ser bastante demandante fazer manualmente a troca de variáveis, pois como vimos, algumas que são significativamente relevante para o modelo, podem deixar de sê-lo na presença de outras.
Para isto, há um algoritmo, chamado stepwise que remove as variáveis que não atingem o nível de significância definido e também já elemina às variáveis com alto nível de multicolinearidade. Vejamos como poderíamos utilizar em nosso modelo que contém a variável “disp”:
modelo_multi_3_step <- step(modelo_multi_3_autocor, k = 3.841459)Start: AIC=73.2
mpg ~ hp + wt + disp
Df Sum of Sq RSS AIC
- disp 1 0.057 195.05 69.365
<none> 194.99 73.197
- hp 1 51.692 246.68 76.880
- wt 1 88.503 283.49 81.331
Step: AIC=69.36
mpg ~ hp + wt
Df Sum of Sq RSS AIC
<none> 195.05 69.365
- hp 1 83.274 278.32 76.900
- wt 1 252.627 447.67 92.109#De onde vem o argumento k = 3.841459? Este é o valor do chi-quadrado para nível de significância de 5% a 1 grau de liberdade. Veja:
qchisq(p = 0.05, df = 1, lower.tail = F)[1] 3.841459round(pchisq(3.841459, df = 1, lower.tail = F),7)[1] 0.05Veja que o procedimento stepwise remove a variável “disp” automaticamente do modelo, gerando um modelo sem a variável com multi-colinearidade.
Apesar da função lm() conseguir lidar com fatores sem a crição de dummies manuais, isso não é verdadeiro para o procedimento stepwise(), portanto, caso utilize oprocedimento stepwise, você deve criar as dummies manualmente, caso contrário a variável não será excluída no stepwise, caso não seja estatisticamente significante.
Tranformações nas variáveis explicativas
Em alguns casos, as variáveis explicativas, também podem ser transformadas de maneira a adequar melhor sua forma funcional. Por exemplo, podemos ter cenários, onde a variável X pode ter uma forma quadrátiva (X^2), logaritmica (log(X)), etc. Nestes casos, podemos lançar mão de estratégias de transformação também para as variáveis explicativas. Vejamos este caso:
Nossa variável hp, é tem sua cauda direita ligeriamente esticada:
df |> ggplot(aes(x=hp))+
geom_histogram(aes(y= after_stat(density)),
colour = 1, fill = "white") +
geom_density() +
labs(title = "Variável hp ANTES da transformação")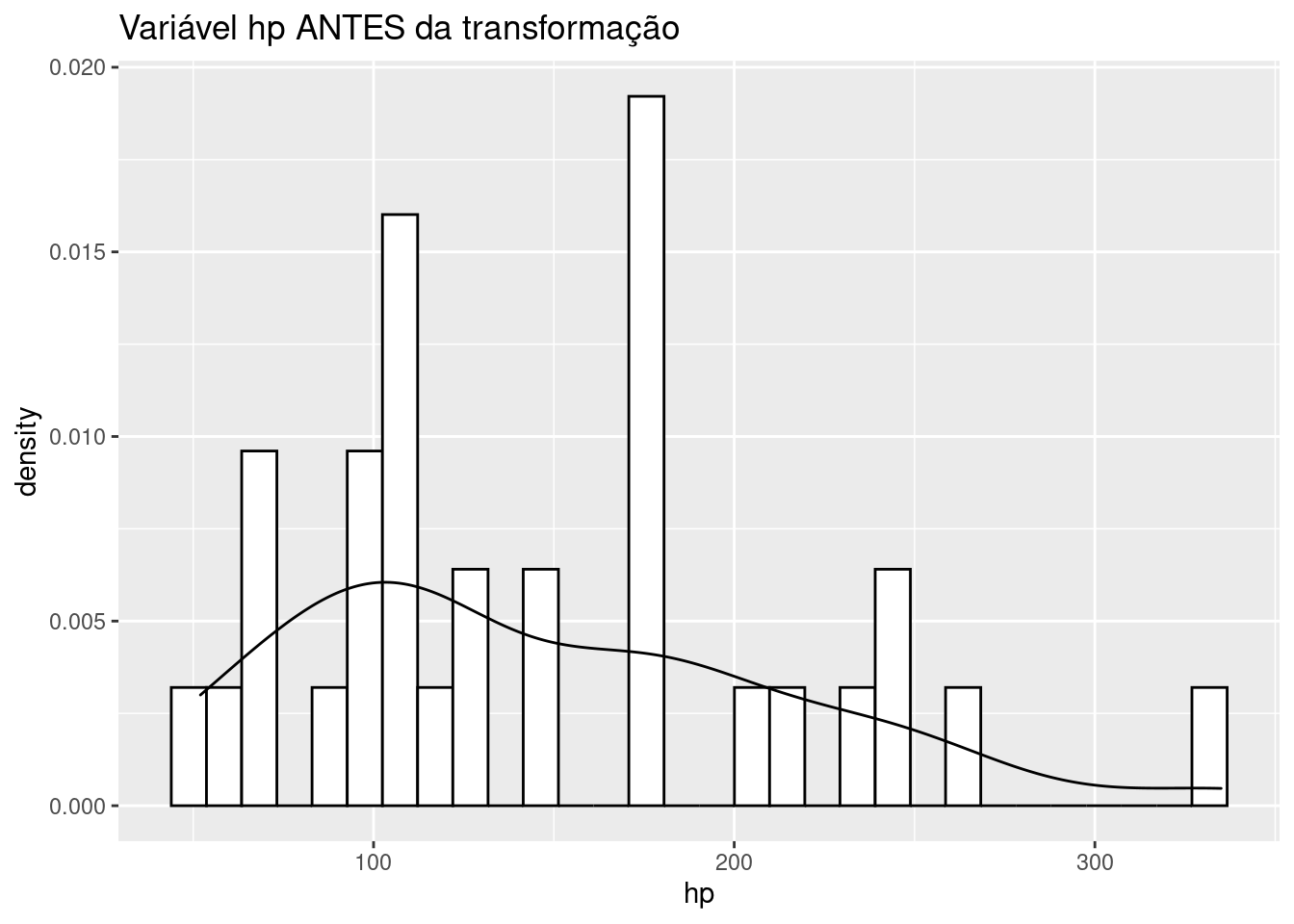
Se fizermos uma transformação logaritmica, podemos deixá-la mais próxima de uma distribuição normal, veja:
df |> ggplot(aes(x=log(hp)))+
geom_histogram(aes(y= after_stat(density)),
colour = 1, fill = "white") +
geom_density()+
labs(title = "Variável hp DEPOIS da transformação")
Não entraremos em detalhes sobre estas transformações neste artigo, mas é importante saber que estas são técnicas muito importantes para melhoria da acurácia de modelos reais.
Vejamos como ficaria um novo modelo, se implementássemos a transformação da variável explicativa (hp) juntamente com o modelo multivariado com transformação de box-cox:
#Transformação Logn da variável hp.
df3 <- df
df3$hp <- log(df3$hp)
#Transformação de Box-Cox
#Estimando o lambda de BoxCox
lambda_BC <- car::powerTransform(df3$mpg)
lambda_BC Estimated transformation parameter
df3$mpg
0.02956537 #Adicionando na base de dados: -->
df3$mpg_bc <- (((df3$mpg ^ lambda_BC$lambda) - 1) / lambda_BC$lambda)
modelo_multi_4_hp_log <- lm(mpg_bc~ hp + wt, df3)
summary(modelo_multi_4_hp_log)
Call:
lm(formula = mpg_bc ~ hp + wt, data = df3)
Residuals:
Min 1Q Median 3Q Max
-0.17830 -0.08425 -0.02470 0.07347 0.28285
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 5.14179 0.24214 21.235 < 2e-16 ***
hp -0.29120 0.06158 -4.729 5.39e-05 ***
wt -0.19524 0.02991 -6.527 3.79e-07 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.1138 on 29 degrees of freedom
Multiple R-squared: 0.8853, Adjusted R-squared: 0.8774
F-statistic: 111.9 on 2 and 29 DF, p-value: 2.306e-14df3$mpg_bc_fitted <- (((modelo_multi_4_hp_log$fitted.values*(lambda_BC$lambda))+
1))^(1/(lambda_BC$lambda))Observe que as variáveis explicativas se mantém relevantes e o R^2 ajustado foi maior que o modelo anterior.
Comparando os modelos
Para comparar os modelos criados até aqui iremos utilizar o R² ajustado:
modelos <- list(uni_hp = modelo_uni,
uni_cyl = modelo_uni_fct,
multi_hp_wt = modelo_multi_1,
multi_hp_wt_bc = modelo_multi_2_bc,
multi_hp_wt_step = modelo_multi_3_step,
multi_hp_wt_log_bc = modelo_multi_4_hp_log)
r2_ajustado <- map_dfr(modelos, ~ summary(.)$adj.r.squared) |>
pivot_longer(cols = everything(), names_to = "modelo", values_to = "R2_ajustado")
r2_ajustadoVisualizando os ajustes dos modelos:
r2_ajustado |>
ggplot(aes(x=as_factor(modelo), y=R2_ajustado, fill = modelo))+
geom_col()+
geom_label(aes(label = round(R2_ajustado,2)), vjust = 0, show.legend = F)+
scale_y_continuous(limits = c(0, 1))+
labs(title = "R2 ajustados dos modelos",x="Modelo",y="R2 Ajustado")+
theme(axis.text.x = element_text(angle = 90))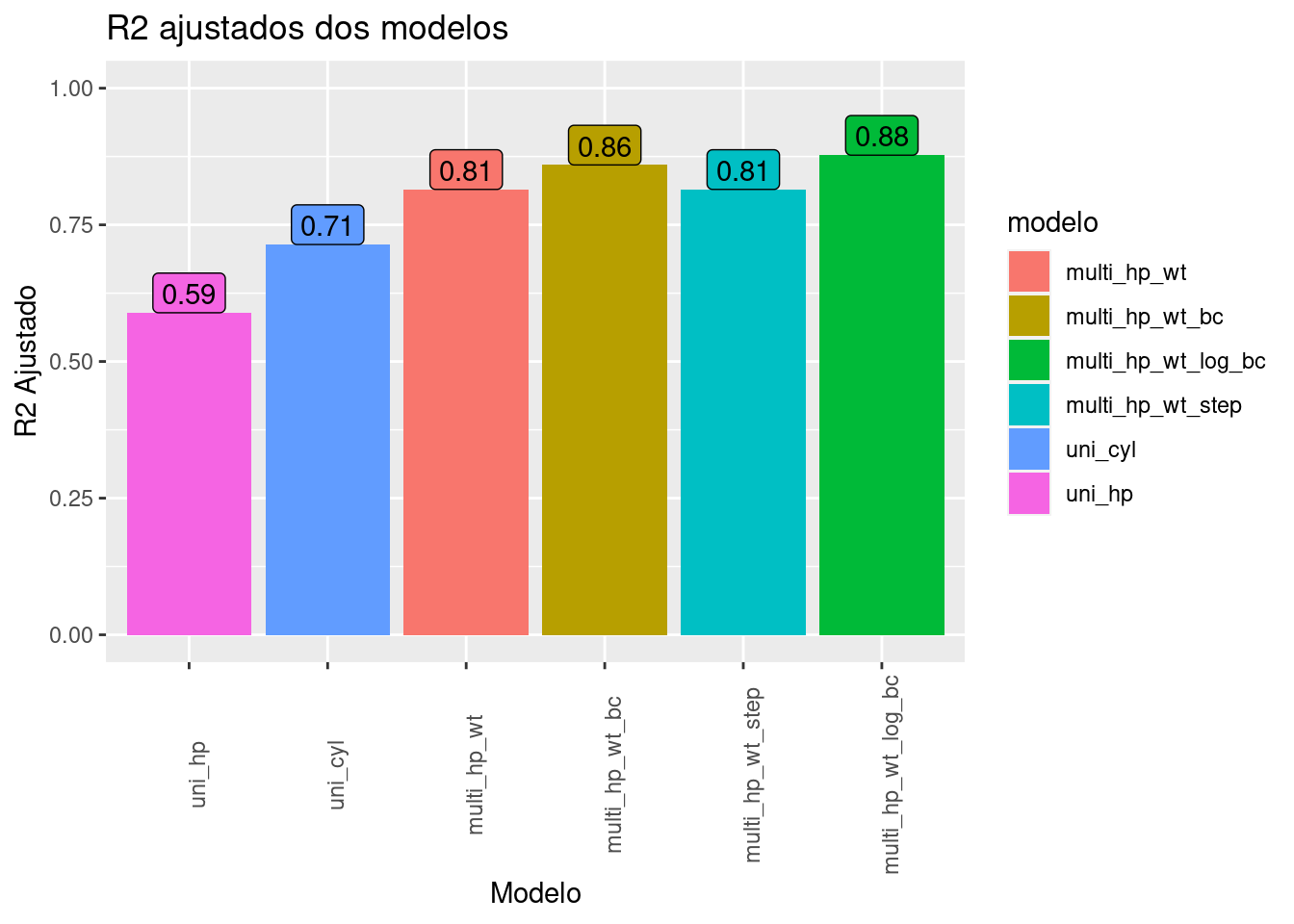
Com isto, temos o modelo multivariado com transformação de Box-Cox na variável dependente e transformação logaritmica na variável explicativa hp com o maior coeficiente de ajuste.
Há diversas outras técnicas de regressão como regressões polinomiais, modelos multi-níveis, etc que podem ser ainda mais adequados para encontrar ajustes melhores do modelo. Apresentamos aqui apenas uma breve introdução sobre o tema.
Modelo Final
Visualizando o modelo com maior R2:
# Ajustando os fatores e junto os fitted values do modelo:
df3_fct <- df3 |> mutate (cyl = as_factor(cyl),
vs = as_factor(vs),
am = as_factor(am),
gear = as_factor(gear),
carb = as_factor(carb))
df3_fct |>
pivot_longer(cols = c(mpg, mpg_bc_fitted),names_to = "tipo", values_to = "mpg") |> group_by(tipo) |>
ggplot(aes(x=name, y=mpg, fill=tipo))+
geom_col(position = position_identity(),
alpha = 0.5)+
theme(axis.text.x = element_text(angle = 90))+
labs(title="Modelo Final Multivariado com \nTransformação de Box-Cox e\nTranformação (log) na variável explicativa hp")+
coord_flip()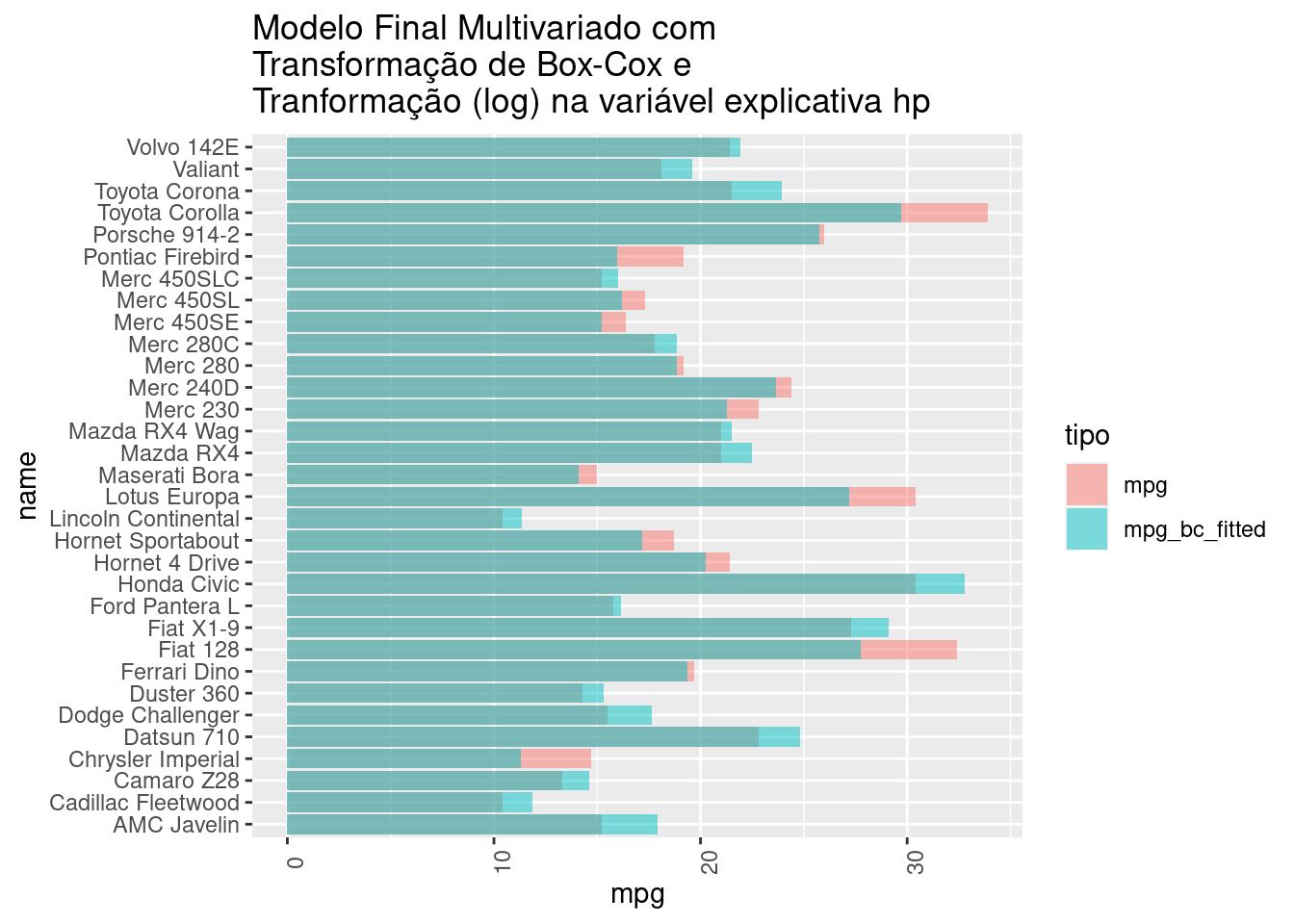
df3_plot <- df3 |>
dplyr::select(model=name, orig = mpg, fitted = mpg_bc_fitted, hp) |>
mutate(hp = exp(hp)) |>
pivot_longer(cols = c("fitted", "orig")) |>
dplyr::rename (categoria = name, mpg = value) |>
mutate (categoria = as_factor(categoria))
df3_plot_sub <- df3_plot |>
filter(categoria =="orig")
df3_plot |>
group_by(categoria) |>
ggplot(aes(x = hp, y = mpg, color = categoria)) +
geom_point(size = 3, alpha = 0.6) +
geom_text_repel(data = df3_plot_sub, aes(x = hp, y = mpg, color = categoria,label=model), color="gray", size=2) +
theme(legend.position = "bottom") +
labs(title="Modelo Final apresentando os \nvalores originais e estimados")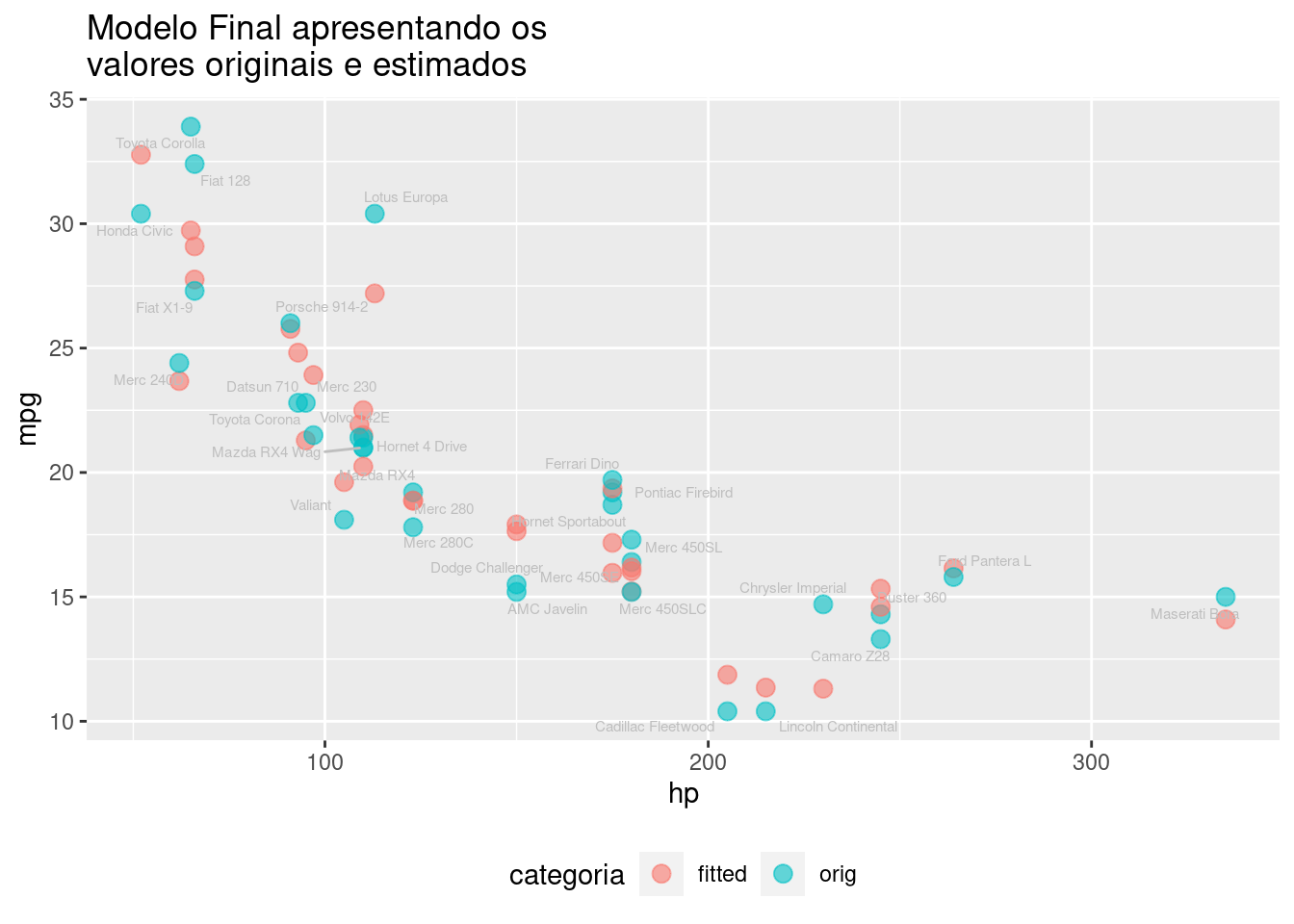
df3_fct |>
ggplot(aes(x = mpg, y = mpg))+
geom_line(linetype = "dashed")+
geom_point(aes(y = mpg_bc_fitted), color = "blue", alpha = 0.6, size = 2)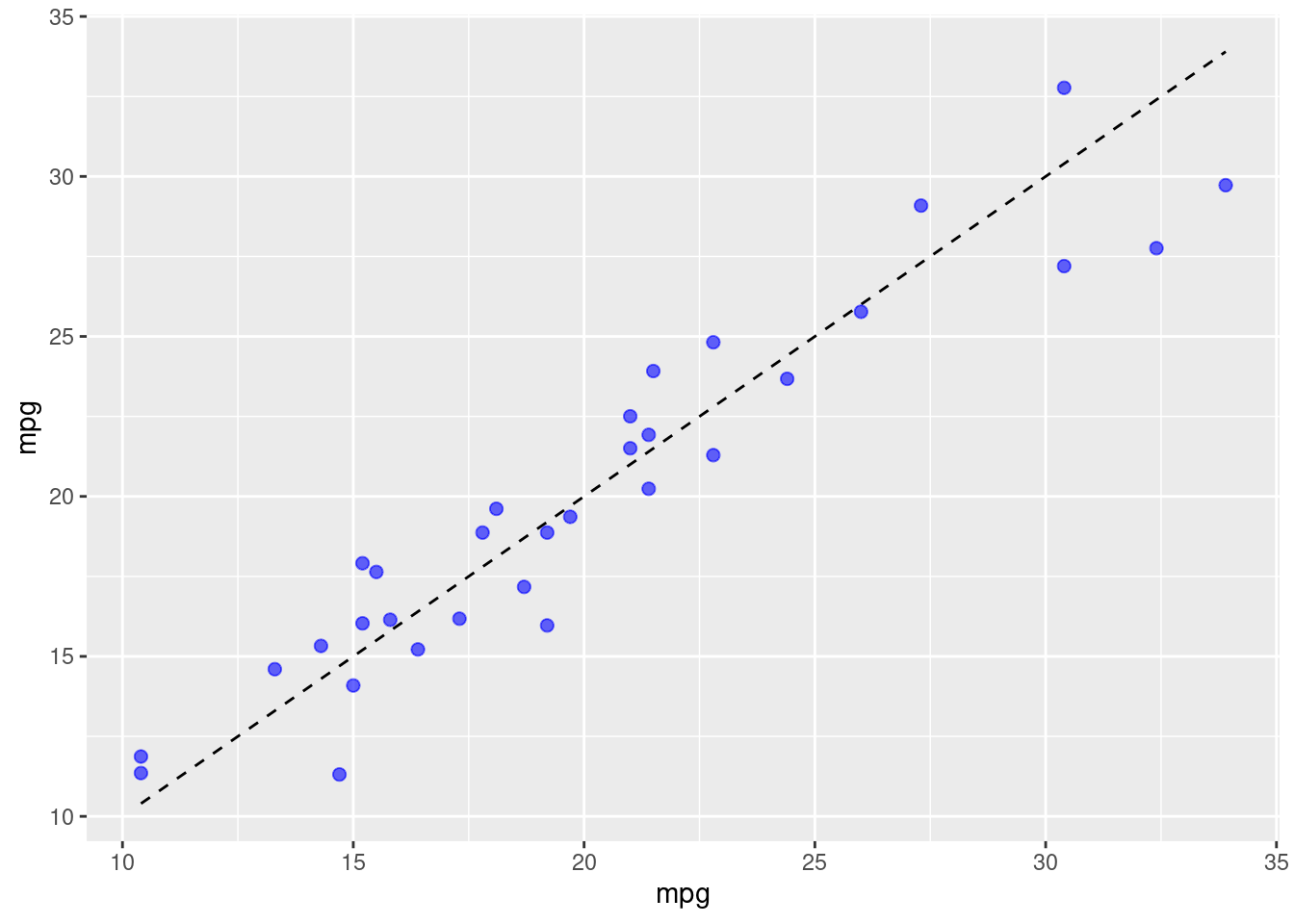
Este modelo seria representado pela seguinte equação:
mpg = ((5.1418 +(-0.2912 *log(hp))+(-0.1952*wt)*0.0296)+1)^{(1/0.02956537)}
Para um veículo com hp = 190 e wt = 2.62, sabendo que o lambda de Box-Cox é de 0.02956537, teríamos:
mpg = (((5.1418 - (0.2912 * log(110)) - (0.1952 * 2.62)) * 0.02956537) + 1)^{(1/0.02956537)}
mpg = 22.5